NGS amplification panel HCV-seq for sequencing hepatitis C virus RNA (Flaviviridae: Hepacivirus)
- Authors: Chanyshev M.D.1, Chernyshova A.S.1,2, Glushchenko A.G.1,2, Grishaeva A.A.1, Makashova V.V.1, Ponezheva Z.B.1, Khafizov K.F.1, Akimkin V.G.1
-
Affiliations:
- Central Research Institute of Epidemiology
- Moscow Institute of Physics and Technology
- Issue: Vol 70, No 4 (2025)
- Pages: 363-373
- Section: ORIGINAL RESEARCHES
- URL: https://journals.rcsi.science/0507-4088/article/view/330075
- DOI: https://doi.org/10.36233/0507-4088-331
- EDN: https://elibrary.ru/fbgobh
- ID: 330075
Cite item
Abstract
Introduction. Hepatitis C is a pressing global public health issue. The high variability of the hepatitis C virus (HCV) complicates its whole-genome sequencing; most studies sequence only specific regions of the genome. There is a need for a simple and reliable method for sequencing the whole genome of HCV.
Objective. Development and validation of NGS panel for whole-genome sequencing of HCV.
Materials and methods. This study presents NGS amplification panel for sequencing the genomes of HCV genotypes 1, 2, and 3. Depending on the genotype, a set of 79, 67, or 89 primers is used. These primers enable amplification of overlapping regions of the HCV genome.
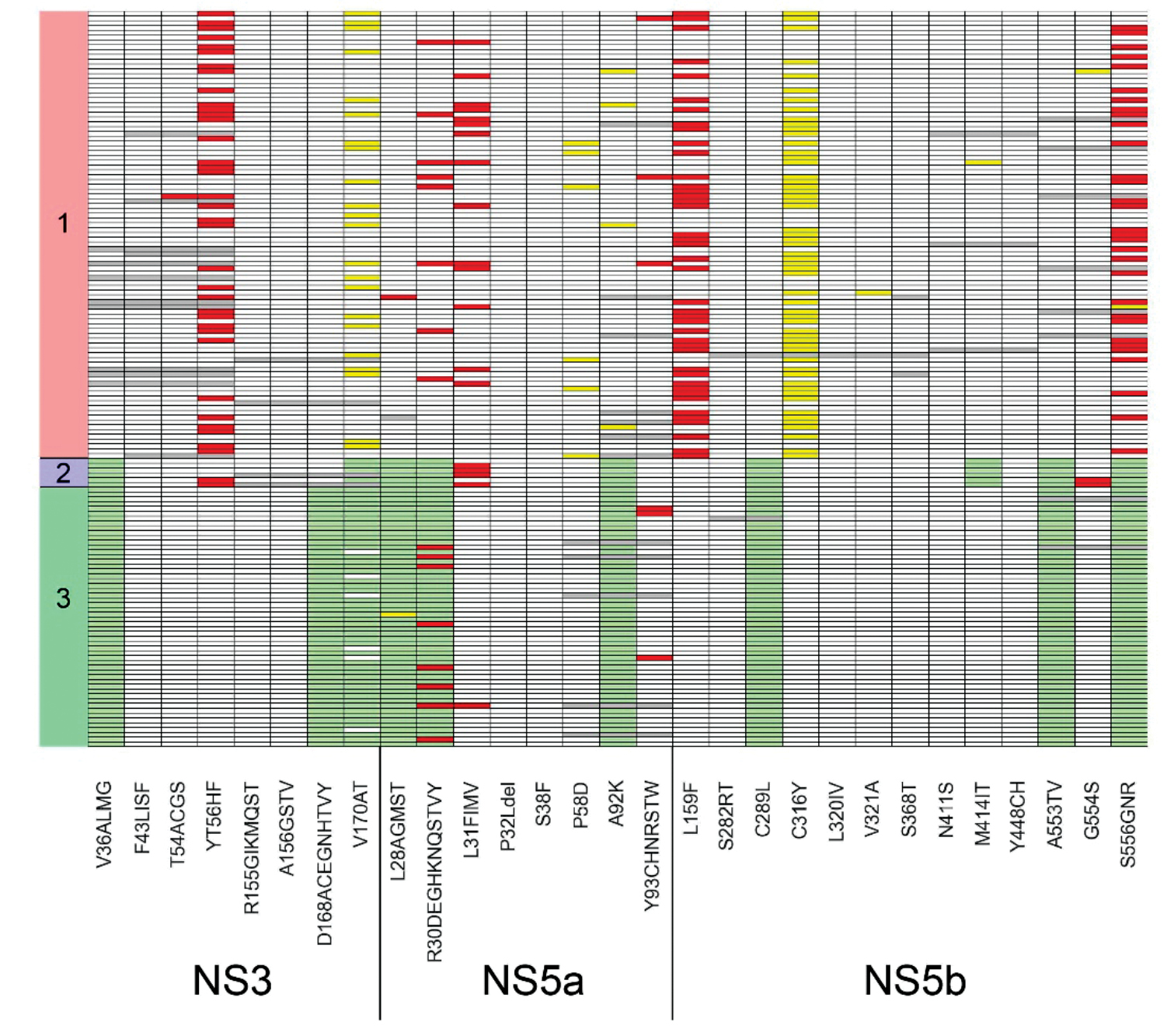
Results. The panel was tested on 153 HCV RNA samples isolated from blood plasma specimens (93/6/54 samples of genotypes 1/2/3, respectively). Shannon entropy analysis showed that genetic heterogeneity within the E2 gene is significantly higher than in other parts of viral genome. The frequency of mutations associated with drug resistance was determined. Specifically, for genotype 1, the following mutation detection rates were observed in NS3: Y56F – 37.6%, V170I – 23.7%; in NS5a: R30Q – 8.6%, P58L/S/T – 6.5%, A92T – 4.3%; in NS5b: L159F – 45.2%, S556G/N – 33.3%.
Conclusion. The current study describes a method for whole-genome sequencing of HCV genotypes 1, 2, and 3. The HCV sequencing panel shows great potential for use in scientific research and epidemiological monitoring.
Keywords
Full Text
##article.viewOnOriginalSite##About the authors
Mikhail D. Chanyshev
Central Research Institute of Epidemiology
Author for correspondence.
Email: chanishq@gmail.com
ORCID iD: 0000-0002-6943-2915
PhD, Senior Researcher, Laboratory for Genomic Research
Russian Federation, 111123, MoscowAnastasia S. Chernyshova
Central Research Institute of Epidemiology; Moscow Institute of Physics and Technology
Email: chernyshova.a@cmd.su
ORCID iD: 0009-0000-2440-1309
Laboratory assistant, Laboratory for Genomic Research, Central Research Institute of Epidemiology
Russian Federation, 111123, Moscow; 141701, DolgoprudnyAlbina G. Glushchenko
Central Research Institute of Epidemiology; Moscow Institute of Physics and Technology
Email: albinagluschenko@gmail.com
ORCID iD: 0009-0002-8851-8703
Laboratory research assistant, Laboratory for Genomic Research, Central Research Institute of Epidemiology
Russian Federation, 111123, Moscow; 141701, DolgoprudnyAntonina A. Grishaeva
Central Research Institute of Epidemiology
Email: antoninagrishaeva@yandex.ru
ORCID iD: 0000-0002-1326-9274
PhD, Researcher of the Clinical Department of Infectious Pathology
Russian Federation, 111123, MoscowVera V. Makashova
Central Research Institute of Epidemiology
Email: veramakashova@yandex.ru
ORCID iD: 0000-0002-0982-3527
Dr. of Sci., Leading Researcher, Clinical Department of Infectious Diseases
Russian Federation, 111123, MoscowZhanna B. Ponezheva
Central Research Institute of Epidemiology
Email: doktorim@mail.ru
ORCID iD: 0000-0002-6539-4878
Dr. of Sci., Associate Professor, Head of the Clinical Department of Infectious Diseases
Russian Federation, 111123, MoscowKamil F. Khafizov
Central Research Institute of Epidemiology
Email: kkhafizov@gmail.com
ORCID iD: 0000-0001-5524-0296
PhD, Head of Laboratory for Genomic Research
Russian Federation, 111123, MoscowVasily G. Akimkin
Central Research Institute of Epidemiology
Email: vgakimkin@yandex.ru
ORCID iD: 0000-0003-4228-9044
Academician of the Russian Academy of Sciences, Dr. of Sci., Professor, Director
Russian Federation, 111123, MoscowReferences
- Isakov V., Nikityuk D. Elimination of HCV in Russia: Barriers and perspective. Viruses. 2022; 14(4): 790. https://doi.org/10.3390/v14040790
- Pimenov N.N., Komarova S.V., Karandashova I.V., Tsapkova N.N., Volchkova E.V., Chulanov V.P. Hepatitis С and its outcomes in Russia: analysis of incidence, prevalence and mortality rates before the start of the programme of infection elimination. Infectious diseases: news, opinions, training. 2018; 16(3): 37–45. https://doi.org/10.20953/1729-9225-2018-3-37-45 https://elibrary.ru/ysjrqt (in Russian)
- Messina J.P., Humphreys I., Flaxman A., Brown A., Cooke G.S., Pybus O.G., et al. Global distribution and prevalence of hepatitis C virus genotypes. Hepatology. 2015; 61(1): 77–87. https://doi.org/10.1002/hep.27259
- Pimenov N., Kostyushev D., Komarova S., Fomicheva A., Urtikov A., Belaia O., et al. Epidemiology and genotype distribution of hepatitis C virus in Russia. Pathogens. 2022; 11(12): 1482. https://doi.org/10.3390/pathogens11121482
- Raimondi S., Bruno S., Mondelli M.U., Maisonneuve P. Hepatitis C virus genotype 1b as a risk factor for hepatocellular carcinoma development: a meta-analysis. J. Hepatol. 2009; 50(6): 1142–54. https://doi.org/10.1016/j.jhep.2009.01.019
- Kalaghatgi P., Sikorski A.M., Knops E., Rupp D., Sierra S. Geno2pheno[HCV] – a Web-based interpretation system to support hepatitis C treatment decisions in the era of direct-acting antiviral agents. PLoS One. 2016; 11(5): e0155869. https://doi.org/10.1371/journal.pone.0155869
- Ahmed A., Felmlee D.J. Mechanisms of hepatitis C viral resistance to direct acting antivirals. Viruses. 2015; 7(12): 6716–29. https://doi.org/10.3390/v7122968
- Yamauchi K., Maekawa S., Osawa L., Komiyama Y., Nakakuki N., Takada H., et al. Single-molecule sequencing of the whole HCV genome revealed envelope deletions in decompensated cirrhosis associated with NS2 and NS5A mutations. J. Gastroenterol. 2024; 59(11): 1021–36. https://doi.org/10.1007/s00535-024-02146-3
- Fishman S.L., Factor S.H., Balestrieri C., Fan X., Dibisceglie A.M., Desai S.M., et al. Mutations in the hepatitis C virus core gene are associated with advanced liver disease and hepatocellular carcinoma. Clin. Cancer Res. 2009; 15(9): 3205–13. https://doi.org/10.1158/1078-0432.ccr-08-2418
- Costantino A., Spada E., Equestre M., Bruni R., Tritarelli E., Coppola N., et al. Naturally occurring mutations associated with resistance to HCV NS5B polymerase and NS3 protease inhibitors in treatment-naïve patients with chronic hepatitis C. Virol. J. 2015; 12: 186. https://doi.org/10.1186/s12985-015-0414-1
- Iio E., Shimada N., Abe H., Atsukawa M., Yoshizawa K., Takaguchi K., et al. Efficacy of daclatasvir/asunaprevir according to resistance-associated variants in chronic hepatitis C with genotype 1. J. Gastroenterol. 2017; 52(1): 94–103. https://doi.org/10.1007/s00535-016-1225-x
- Spitz N., Barros J.J., do Ó K.M., Brandão-Mello C.E., Araujo N.M. The first complete genome sequences of hepatitis C virus subtype 2b from Latin America: molecular characterization and phylogeographic analysis. Viruses. 2019; 11(11): 1000. https://doi.org/10.3390/v11111000
- Bull R.A., Eltahla A.A., Rodrigo C., Koekkoek S.M., Walker M., Pirozyan M.R., et al. A method for near full-length amplification and sequencing for six hepatitis C virus genotypes. BMC Genomics. 2016; 17: 247. https://doi.org/10.1186/s12864-016-2575-8
- Aisyah D.N., Story A., Kremyda-Vlachou M., Kozlakidis Z., Shalcross L., Hayward A. Assessing hepatitis C virus distribution among vulnerable populations in London using whole genome sequencing: results from the TB-REACH study. WellcomeOpenRes. 2021; 6: 229. https://doi.org/10.12688/wellcomeopenres.16907.1
- Kotova V.O., Balakhontseva L.A., Bazykina E.A., Trotsenko O.E., Beldy V.N., Kirdyashova S.E. Genetic diversity of hepatitis c virus in Nanaian region, Khabarovsk territory. Infektsiya i immunitet. 2021; 11(1): 148–56. https://doi.org/10.15789/2220-7619-GDO-1265 https://elibrary.ru/enfery (in Russian)
- Reingardt D.E., Ostankova Yu.V., Lyalina L.V., Anufrieva E.V., Semenov A.V., Totolian A.A. Distribution of hepatitis С virus drug resistance mutations among patients with recurrence of the disease during therapy with direct antiviral drugs. VICh-infektsiya i immunosupressii. 2023; 15(4): 86–93. https://doi.org/10.22328/2077-9828-2023-15-4-86-93 (in Russian)
- Isakov V., Hedskog C., Wertheim J.O., Hostager R.E., Parhy B., Schneider A.B., et al. Prevalence of resistance-associated substitutions and phylogenetic analysis of hepatitis C virus infection in Russia. Int. J. Infect. Dis. 2021; 113: 36–42. https://doi.org/10.1016/j.ijid.2021.09.041
- Chanyshev M.D., Vlasenko N.V., Roev G.V., Kotov I.A., Glushchenko A.G., Makashova V.V., et al. NGS amplification panel for HBV (Hepadnaviridae: Orthohepadnavirus) sequencing. Voprosy virusologii. 2024; 69(1): 65–75. https://doi.org/10.36233/0507-4088-212 (in Russian)
- Cheng X.D., Xu H.F., Wei X.M., Zhou H.Z. Variation analysis of E1 and E2 in HCV subtypes. Arch. Virol. 2015; 160(10): 2479–82. https://doi.org/10.1007/s00705-015-2533-9
- Kandel S., Hartzell S.L., Ingold A.K., Turner G.A., Kennedy J.L., Ussery D.W. Genomic surveillance of SARS-CoV-2 using long-range PCR primers. Front. Microbiol. 2024; 15: 1272972. https://doi.org/10.3389/fmicb.2024.1272972
- Koskela von Sydow A., Lindqvist C.M., Asghar N., Johansson M., Sundqvist M., Mölling P., et al. Comparison of SARS-CoV-2 whole genome sequencing using tiled amplicon enrichment and bait hybridization. Sci. Rep. 2023; 13(1): 6461. https://doi.org/10.1038/s41598-023-33168-1
- Yoshimi S., Imamura M., Murakami E., Hiraga N., Tsuge M., Kawakami Y., et al. Long term persistence of NS5A inhibitor-resistant hepatitis C virus in patients who failed daclatasvir and asunaprevir therapy. J. Med. Virol. 2015; 87(11): 1913–20. https://doi.org/10.1002/jmv.24255
- Wyles D.L., Luetkemeyer A.F. Understanding hepatitis C virus drug resistance: clinical implications for current and future regimens. Top Antivir. Med. 2017; 25(3): 103–9.
Supplementary files