Амплификационная панель NGS HCV-seq для таргетного секвенирования РНК вируса гепатита С (Flaviviridae: Hepacivirus)
- Авторы: Чанышев М.Д.1, Чернышова А.С.1,2, Глущенко А.Г.1,2, Гришаева А.А.1, Макашова В.В.1, Понежева Ж.Б.1, Хафизов К.Ф.1, Акимкин В.Г.1
-
Учреждения:
- ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора
- ФГАОУ ВО «Московский физико-технический институт (национальный исследовательский университет)»
- Выпуск: Том 70, № 4 (2025)
- Страницы: 363-373
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://journals.rcsi.science/0507-4088/article/view/330075
- DOI: https://doi.org/10.36233/0507-4088-331
- EDN: https://elibrary.ru/fbgobh
- ID: 330075
Цитировать
Аннотация
Введение. Гепатит С является актуальной проблемой здравоохранения во всем мире. Высокая вариабельность вируса гепатита С (ВГС) осложняет его полногеномное секвенирование, в большинстве исследований секвенированы только отдельные регионы генома. Существует необходимость в простом и надежном методе секвенирования полного генома ВГС.
Цель работы. Создание и апробация панели NGS для полногеномного секвенирования ВГС.
Материалы и методы. В работе представлена амплификационная панель NGS для секвенирования генома ВГС генотипов 1, 2 и 3. В зависимости от генотипа использован набор, состоящий из 79, 67 или 89 праймеров, при помощи которых осуществляется амплификация перекрывающихся участков генома ВГС.
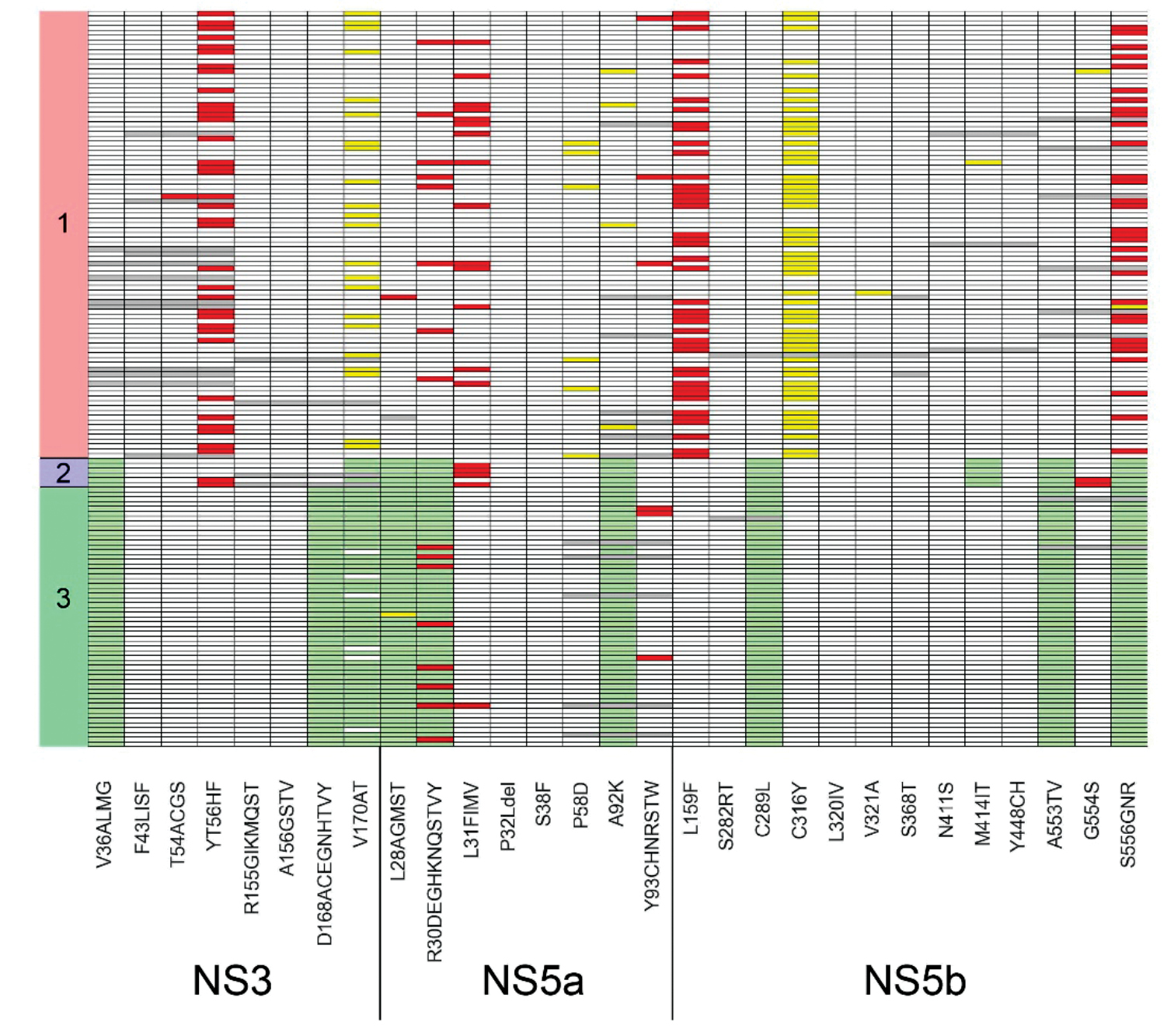
Результаты. Панель была апробирована на 153 образцах РНК ВГC, выделенных из крови (93/6/54 образца генотипов 1/2/3 соответственно). Анализ энтропии Шеннона показал, что в пределах гена E2 генетическая неоднородность значительно выше, чем в остальной части генома. Была определена частота встречаемости мутаций, ассоциированных с лекарственной резистентностью. В частности, для генотипа 1 была отмечена следующая частота встречаемости мутаций в NS3: Y56F – 37,6%, V170I – 23,7%; в NS5a: R30Q – 8,6%, P58L/S/T – 6,5%, A92T – 4,3%; в NS5b: L159F – 45,2%, S556G/N – 33,3%.
Заключение. В работе изложен способ полногеномного секвенирования генома ВГС генотипов 1, 2 и 3. Панель для секвенирования ВГС обладает большим потенциалом для использования в научных исследованиях и эпидемиологическом мониторинге.
Ключевые слова
Полный текст
Открыть статью на сайте журналаОб авторах
Михаил Дамирович Чанышев
ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора
Автор, ответственный за переписку.
Email: chanishq@gmail.com
ORCID iD: 0000-0002-6943-2915
канд. биол. наук, старший научный сотрудник Лаборатории геномных исследований
Россия, 111123, г. МоскваАнастасия Сергеевна Чернышова
ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора; ФГАОУ ВО «Московский физико-технический институт (национальный исследовательский университет)»
Email: chernyshova.a@cmd.su
ORCID iD: 0009-0000-2440-1309
лаборант Лаборатории геномных исследований ЦНИИ Эпидемиологии Роспотребнадзора
Россия, 111123, г. Москва; 141701, г. ДолгопрудныйАльбина Григорьевна Глущенко
ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора; ФГАОУ ВО «Московский физико-технический институт (национальный исследовательский университет)»
Email: albinagluschenko@gmail.com
ORCID iD: 0009-0002-8851-8703
лаборант-исследователь Лаборатории геномных исследований ЦНИИ Эпидемиологии Роспотребнадзора
Россия, 111123, г. Москва; 141701, г. ДолгопрудныйАнтонина Алексеевна Гришаева
ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора
Email: antoninagrishaeva@yandex.ru
ORCID iD: 0000-0002-1326-9274
канд. мед. наук, научный сотрудник Клинического отдела инфекционной патологии
Россия, 111123, г. МоскваВера Васильевна Макашова
ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора
Email: veramakashova@yandex.ru
ORCID iD: 0000-0002-0982-3527
д-р мед. наук, ведущий научный сотрудник Клинического отдела инфекционной патологии
Россия, 111123, г. МоскваЖанна Бетовна Понежева
ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора
Email: doktorim@mail.ru
ORCID iD: 0000-0002-6539-4878
д-р мед. наук, доцент, заведующая Клиническим отделом инфекционной патологии
Россия, 111123, г. МоскваКамиль Фаридович Хафизов
ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора
Email: kkhafizov@gmail.com
ORCID iD: 0000-0001-5524-0296
канд. биол. наук, заведующий Лабораторией геномных исследований
Россия, 111123, г. МоскваВасилий Геннадьевич Акимкин
ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора
Email: vgakimkin@yandex.ru
ORCID iD: 0000-0003-4228-9044
академик РАН, доктор медицинских наук, профессор, директор
Россия, 111123, г. МоскваСписок литературы
- Isakov V., Nikityuk D. Elimination of HCV in Russia: Barriers and perspective. Viruses. 2022; 14(4): 790. https://doi.org/10.3390/v14040790
- Пименов Н.Н., Комарова С.В., Карандашова И.В., Цапкова Н.Н., Волчкова Е.В., Чуланов В.П. Гепатит С и его исходы в России: анализ заболеваемости, распространенности и смертности до начала программы ликвидации инфекции. Инфекционные болезни. 2018; 16(3): 37–45. https://doi.org/10.20953/1729-9225-2018-3-37-45 https://elibrary.ru/ysjrqt
- Messina J.P., Humphreys I., Flaxman A., Brown A., Cooke G.S., Pybus O.G., et al. Global distribution and prevalence of hepatitis C virus genotypes. Hepatology. 2015; 61(1): 77–87. https://doi.org/10.1002/hep.27259
- Pimenov N., Kostyushev D., Komarova S., Fomicheva A., Urtikov A., Belaia O., et al. Epidemiology and genotype distribution of hepatitis C virus in Russia. Pathogens. 2022; 11(12): 1482. https://doi.org/10.3390/pathogens11121482
- Raimondi S., Bruno S., Mondelli M.U., Maisonneuve P. Hepatitis C virus genotype 1b as a risk factor for hepatocellular carcinoma development: a meta-analysis. J. Hepatol. 2009; 50(6): 1142–54. https://doi.org/10.1016/j.jhep.2009.01.019
- Kalaghatgi P., Sikorski A.M., Knops E., Rupp D., Sierra S. Geno2pheno[HCV] – a Web-based interpretation system to support hepatitis C treatment decisions in the era of direct-acting antiviral agents. PLoS One. 2016; 11(5): e0155869. https://doi.org/10.1371/journal.pone.0155869
- Ahmed A., Felmlee D.J. Mechanisms of hepatitis C viral resistance to direct acting antivirals. Viruses. 2015; 7(12): 6716–29. https://doi.org/10.3390/v7122968
- Yamauchi K., Maekawa S., Osawa L., Komiyama Y., Nakakuki N., Takada H., et al. Single-molecule sequencing of the whole HCV genome revealed envelope deletions in decompensated cirrhosis associated with NS2 and NS5A mutations. J. Gastroenterol. 2024; 59(11): 1021–36. https://doi.org/10.1007/s00535-024-02146-3
- Fishman S.L., Factor S.H., Balestrieri C., Fan X., Dibisceglie A.M., Desai S.M., et al. Mutations in the hepatitis C virus core gene are associated with advanced liver disease and hepatocellular carcinoma. Clin. Cancer Res. 2009; 15(9): 3205–13. https://doi.org/10.1158/1078-0432.ccr-08-2418
- Costantino A., Spada E., Equestre M., Bruni R., Tritarelli E., Coppola N., et al. Naturally occurring mutations associated with resistance to HCV NS5B polymerase and NS3 protease inhibitors in treatment-naïve patients with chronic hepatitis C. Virol. J. 2015; 12: 186. https://doi.org/10.1186/s12985-015-0414-1
- Iio E., Shimada N., Abe H., Atsukawa M., Yoshizawa K., Takaguchi K., et al. Efficacy of daclatasvir/asunaprevir according to resistance-associated variants in chronic hepatitis C with genotype 1. J. Gastroenterol. 2017; 52(1): 94–103. https://doi.org/10.1007/s00535-016-1225-x
- Spitz N., Barros J.J., do Ó K.M., Brandão-Mello C.E., Araujo N.M. The first complete genome sequences of hepatitis C virus subtype 2b from Latin America: molecular characterization and phylogeographic analysis. Viruses. 2019; 11(11): 1000. https://doi.org/10.3390/v11111000
- Bull R.A., Eltahla A.A., Rodrigo C., Koekkoek S.M., Walker M., Pirozyan M.R., et al. A method for near full-length amplification and sequencing for six hepatitis C virus genotypes. BMC Genomics. 2016; 17: 247. https://doi.org/10.1186/s12864-016-2575-8
- Aisyah D.N., Story A., Kremyda-Vlachou M., Kozlakidis Z., Shalcross L., Hayward A. Assessing hepatitis C virus distribution among vulnerable populations in London using whole genome sequencing: results from the TB-REACH study. WellcomeOpenRes. 2021; 6: 229. https://doi.org/10.12688/wellcomeopenres.16907.1
- Котова В.О., Балахонцева Л.А., Базыкина Е.А., Троценко О.Е., Бельды В.Н., Кирдяшова С.Е. Генетическое разнообразие вируса гепатита С среди населения Нанайского района Хабаровского края. Инфекция и иммунитет. 2021; 11(1): 148–56. https://doi.org/10.15789/2220-7619-GDO-1265 https://elibrary.ru/enfery
- Рейнгардт Д.Э., Останкова Ю.В., Лялина Л.В., Ануфриева Е.В., Семенов А.В., Тотолян А.А. Распространенность мутаций лекарственной устойчивости вируса гепатита С среди пациентов с рецидивом заболевания на терапии препаратами прямого противовирусного действия. ВИЧ-инфекция и иммуносупрессии. 2023; 15(4): 86–93. https://doi.org/10.22328/2077-9828-2023-15-4-86-93
- Isakov V., Hedskog C., Wertheim J.O., Hostager R.E., Parhy B., Schneider A.B., et al. Prevalence of resistance-associated substitutions and phylogenetic analysis of hepatitis C virus infection in Russia. Int. J. Infect. Dis. 2021; 113: 36–42. https://doi.org/10.1016/j.ijid.2021.09.041
- Чанышев М.Д., Власенко Н.В., Роев Г.В., Котов И.А., Глущенко А.Г., Макашова В.В. и др. Амплификационная панель NGS для секвенирования ДНК вируса гепатита В (Hepadnaviridae: Orthohepa DNA virus). Вопросы вирусологии. 2024; 69(1): 65–75. https://doi.org/10.36233/0507-4088-212
- Cheng X.D., Xu H.F., Wei X.M., Zhou H.Z. Variation analysis of E1 and E2 in HCV subtypes. Arch. Virol. 2015; 160(10): 2479–82. https://doi.org/10.1007/s00705-015-2533-9
- Kandel S., Hartzell S.L., Ingold A.K., Turner G.A., Kennedy J.L., Ussery D.W. Genomic surveillance of SARS-CoV-2 using long-range PCR primers. Front. Microbiol. 2024; 15: 1272972. https://doi.org/10.3389/fmicb.2024.1272972
- Koskela von Sydow A., Lindqvist C.M., Asghar N., Johansson M., Sundqvist M., Mölling P., et al. Comparison of SARS-CoV-2 whole genome sequencing using tiled amplicon enrichment and bait hybridization. Sci. Rep. 2023; 13(1): 6461. https://doi.org/10.1038/s41598-023-33168-1
- Yoshimi S., Imamura M., Murakami E., Hiraga N., Tsuge M., Kawakami Y., et al. Long term persistence of NS5A inhibitor-resistant hepatitis C virus in patients who failed daclatasvir and asunaprevir therapy. J. Med. Virol. 2015; 87(11): 1913–20. https://doi.org/10.1002/jmv.24255
- Wyles D.L., Luetkemeyer A.F. Understanding hepatitis C virus drug resistance: clinical implications for current and future regimens. Top Antivir. Med. 2017; 25(3): 103–9.
Дополнительные файлы