Genetic characteristics of the isolate of human adenovirus type 55 (Adenoviridae: Mastadenovirus) isolated in Moscow in 2022
- 作者: Shein D.A.1, Ryzhova N.N.1, Kunda M.S.1, Ermolova E.I.1, Ozharovskaia T.A.1, Popova O.1, Nikitenko N.A.1, Krasnoslobodtsev K.G.1, Burtseva E.I.1, Zubkova O.V.1, Voronina O.L.1, Gintsburg A.L.1
-
隶属关系:
- National Research Center for Epidemiology and Microbiology named after Honorary Academician N.F. Gamaleya of the Ministry of Health of the Russian Federation
- 期: 卷 70, 编号 5 (2025)
- 页面: 431-443
- 栏目: ORIGINAL RESEARCHES
- URL: https://journals.rcsi.science/0507-4088/article/view/353626
- DOI: https://doi.org/10.36233/0507-4088-297
- EDN: https://elibrary.ru/aatdbs
- ID: 353626
如何引用文章
详细
Introduction. Adenovirus infection occurs globally in the form of sporadic cases and isolated outbreaks. Human adenovirus type 55 (HAdV-55), endemic in China and South Korea, causes acute respiratory viral infections (ARVI) of varying severity, both among the civilian population and in military units in different countries of the world. Genomic research facilitates reliable identification of HAdV-55.
The aim of this study was to identify HAdV isolated in Moscow in 2022, as well as to conduct whole-genome sequencing and comparative genomic research.
Materials and methods. HAdV-55 was isolated from a sample of a patient hospitalized with pneumonia and analyzed using restriction fragment length polymorphism analysis and whole-genome sequencing. Bioinformatics comparative analysis was performed on a sample of sequences of 83 isolates.
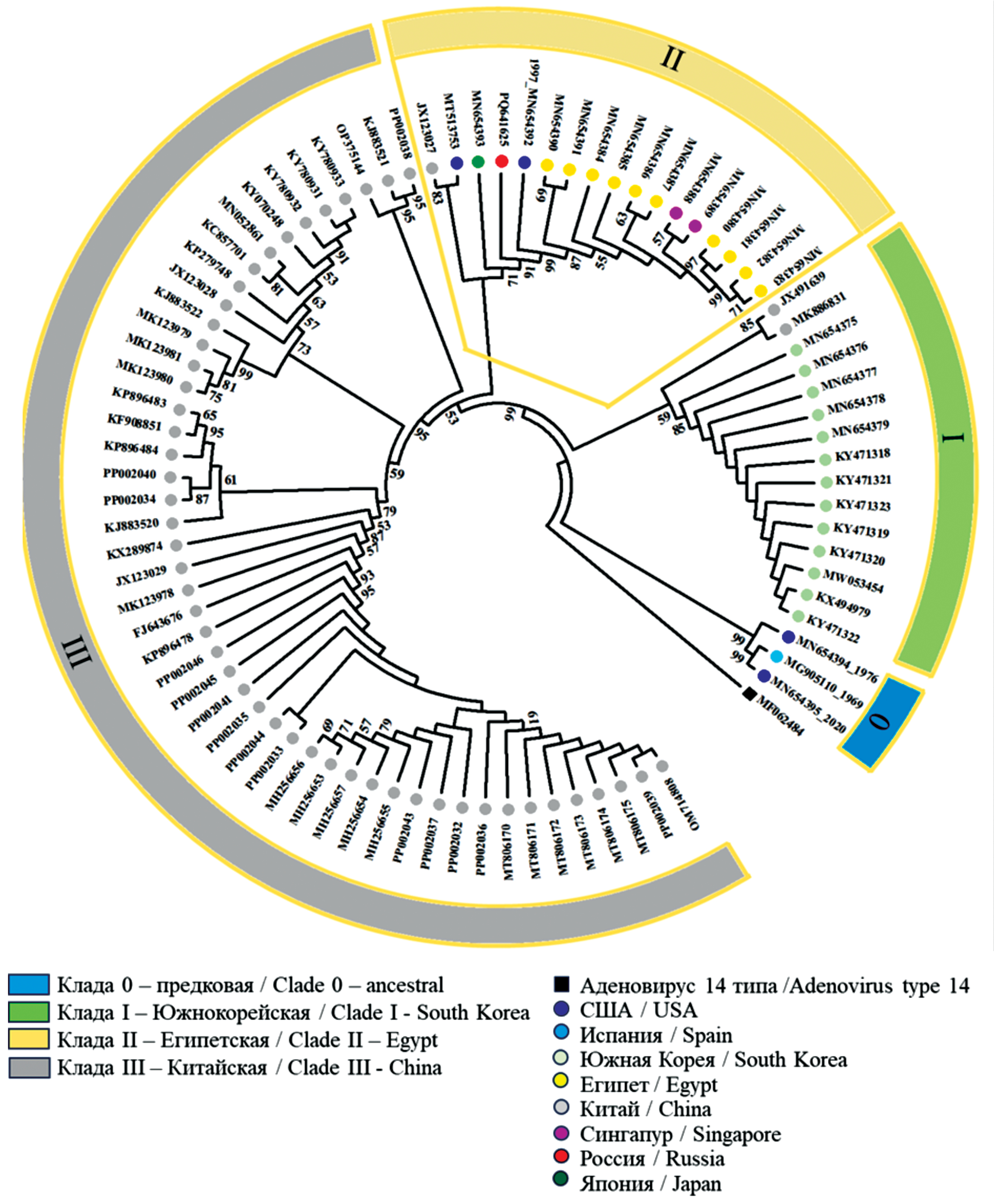
Results. The whole-genome sequencing of first isolated in Russia HAdV-55 was conducted. The sequence of isolate SCV3008:Ad55 was deposited in GenBank (Accession Number PQ641625). Unique mutations in the SCV3008:Ad55 genome were identified, one of which resulted in a conservative T29A substitution in the penton that did not affect its functions. Phylogenetic analysis showed clustering of SCV3008:Ad55 with isolates of clade II, which included representatives of 7 countries on different continents, indicating a wide distribution of HAdV-55. Isolates from endemic regions of China and South Korea formed separate clades. The study of microsatellite length polymorphism in untranslated regions of the genome became an additional tool for distinguishing closely related genomes.
Conclusion. The obtained genomic information laid the foundation for further monitoring for HAdV-55 in Russia and demonstrated the informativeness and significance of whole-genome studies for monitoring adenoviruses. The development and implementation of genotyping methods aimed at detecting HAdV-55 and other clinically relevant genotypes will significantly improve the effectiveness of the diagnosis of adenovirus infections with the threat of developing bronchopneumonia.
作者简介
Daniil Shein
National Research Center for Epidemiology and Microbiology named after Honorary Academician N.F. Gamaleya of the Ministry of Health of the Russian Federation
Email: daniil.schein@yandex.ru
ORCID iD: 0009-0003-3768-9817
Postgraduate Student, Laboratory of genome analysis
俄罗斯联邦, Moscow, 123098Natalya Ryzhova
National Research Center for Epidemiology and Microbiology named after Honorary Academician N.F. Gamaleya of the Ministry of Health of the Russian Federation
Email: rynatalia@yandex.ru
ORCID iD: 0000-0001-5361-870X
Cand. Sci. (Biol.), Senior Researcher, Laboratory of genome analysis
俄罗斯联邦, Moscow, 123098Marina Kunda
National Research Center for Epidemiology and Microbiology named after Honorary Academician N.F. Gamaleya of the Ministry of Health of the Russian Federation
Email: markunda99@gmail.com
ORCID iD: 0000-0003-1945-0397
Cand. Sci. (Biol.), Senior Researcher, Laboratory of genome analysis
俄罗斯联邦, Moscow, 123098Ekaterina Ermolova
National Research Center for Epidemiology and Microbiology named after Honorary Academician N.F. Gamaleya of the Ministry of Health of the Russian Federation
Email: aksenova16@yandex.ru
ORCID iD: 0000-0002-0437-9404
Cand. Sci. (Biol.), Senior Researcher, Laboratory of genome analysis
俄罗斯联邦, Moscow, 123098Tatiana Ozharovskaia
National Research Center for Epidemiology and Microbiology named after Honorary Academician N.F. Gamaleya of the Ministry of Health of the Russian Federation
Email: t.ozh@yandex.ru
ORCID iD: 0000-0001-7147-1553
Cand. Sci. (Biol.), Senior Researcher, Immunobiotechnology laboratory
俄罗斯联邦, Moscow, 123098Olga Popova
National Research Center for Epidemiology and Microbiology named after Honorary Academician N.F. Gamaleya of the Ministry of Health of the Russian Federation
Email: olga.popova31@yandex.ru
ORCID iD: 0000-0003-3248-1227
junior researcher, Immunobiotechnology laboratory
俄罗斯联邦, Moscow, 123098Natalia Nikitenko
National Research Center for Epidemiology and Microbiology named after Honorary Academician N.F. Gamaleya of the Ministry of Health of the Russian Federation
Email: nan-nikitenko@yandex.ru
ORCID iD: 0000-0001-5829-744X
Cand. Sci. (Biol.), Senior Researcher, Head of Medical Department
俄罗斯联邦, Moscow, 123098Kirill Krasnoslobodtsev
National Research Center for Epidemiology and Microbiology named after Honorary Academician N.F. Gamaleya of the Ministry of Health of the Russian Federation
Email: kkg_87@mail.ru
ORCID iD: 0000-0003-1745-9128
researcher, Influenza etiology and epidemiology laboratory
俄罗斯联邦, Moscow, 123098Elena Burtseva
National Research Center for Epidemiology and Microbiology named after Honorary Academician N.F. Gamaleya of the Ministry of Health of the Russian Federation
Email: elena-burtseva@yandex.ru
ORCID iD: 0000-0003-2518-6801
Dr. Sci. (Medicine), Head, Influenza etiology and epidemiology laboratory
俄罗斯联邦, Moscow, 123098Olga Zubkova
National Research Center for Epidemiology and Microbiology named after Honorary Academician N.F. Gamaleya of the Ministry of Health of the Russian Federation
编辑信件的主要联系方式.
Email: olga-zubkova@yandex.ru
ORCID iD: 0000-0001-7893-8419
Cand. Sci. (Biol.), Leading Researcher, Immunobiotechnology laboratory
俄罗斯联邦, Moscow, 123098Olga Voronina
National Research Center for Epidemiology and Microbiology named after Honorary Academician N.F. Gamaleya of the Ministry of Health of the Russian Federation
Email: olv550@gmail.com
ORCID iD: 0000-0001-7206-3594
Cand. Sci. (Biol.), Assistant Professor, Leading Researcher, Head of Laboratory of genome analysis
俄罗斯联邦, Moscow, 123098Alexander Gintsburg
National Research Center for Epidemiology and Microbiology named after Honorary Academician N.F. Gamaleya of the Ministry of Health of the Russian Federation
Email: gintsburg@gamaleya.org
ORCID iD: 0000-0003-1769-5059
Dr. Sci. (Biol.), RAS academician, professor, Director
俄罗斯联邦, Moscow, 123098参考
- Lynch J.P. 3rd, Kajon A.E. Adenovirus: epidemiology, global spread of novel serotypes, and advances in treatment and prevention. Semin. Respir. Crit. Care Med. 2016; 37(4): 586–602. https://doi.org/10.1055/s-0036-1584923
- Coleman K.K., Wong C.C., Jayakumar J., Nguyen T.T., Wong A.W.L., Yadana S., et al. Adenoviral infections in Singapore: Should new antiviral therapies and vaccines be adopted? J. Infect. Dis. 2020; 221(4): 566–77. https://doi.org/10.1093/infdis/jiz489
- Xu W., Xu Z., Huang L., Qin E.Q., Zhang J.L., Zhao P., et al. Transcriptome sequencing identifies novel immune response genes highly related to the severity of human adenovirus type 55 infection. Front. Microbiol. 2019; 10: 130. https://doi.org/10.3389/fmicb.2019.00130
- Kajon A.E., Lamson D.M., St. George K. Emergence and re-emergence of respiratory adenoviruses in the United States. Curr. Opin. Virol. 2019; 34: 63–9. https://doi.org/10.1016/j.coviro.2018.12.004
- Dhingra A., Hage E., Ganzenmueller T., Böttcher S., Hofmann J., Hamprecht K., et al. Molecular Evolution of Human Adenovirus (HAdV) Species C. Sci Rep. 2019; 9(1): 1039. https://doi.org/10.1038/s41598-018-37249-4
- Scott M.K., Chommanard C., Lu X., Appelgate D., Grenz L., Schneider E., et al. Human adenovirus associated with severe respiratory infection, Oregon, USA, 2013–2014. Emerg. Infect. Dis. 2016; 22(6): 1044–51. https://doi.org/10.3201/eid2206.151898
- Hierholzer J.C., Pumarola A., Rodriguez-Torres A., Beltran M. Occurrence of respiratory illness due to an atypical strain of adenovirus type 11 during a large outbreak in Spanish military recruits. Am. J. Epidemiol. 1974; 99(6): 434–42. https://doi.org/10.1093/oxfordjournals.aje.a121632
- Li Q.G., Hambraeus J., Wadell G. Genetic relationship between thirteen genome types of adenovirus 11, 34, and 35 with different tropisms. Intervirology. 1991; 32(6): 338–50. https://doi.org/10.1159/000150218
- Yang Z., Zhu Z., Tang L., Wang L., Tan X., Yu P., et al. Genomic analyses of recombinant adenovirus type 11a in China. J. Clin. Microbiol. 2009; 47(10): 3082–90. https://doi.org/10.1128/JCM.00282-09
- Seto D., Chodosh J., Brister J.R., Jones M.S. Using the whole-genome sequence to characterize and name human adenoviruses. J. Virol. 2011; 85(11): 5701–2. https://doi.org/10.1128/JVI.00354-11
- Seto D., Jones M.S., Dyer D.W., Chodosh J. Characterizing, typing, and naming human adenovirus type 55 in the era of whole genome data. J. Clin. Virol. 2013; 58(4): 741–2. https://doi.org/10.1016/j.jcv.2013.09.025
- Centers for Disease Control and Prevention (CDC). Civilian outbreak of adenovirus acute respiratory disease – South Dakota, 1997. MMWR Morb. Mortal. Wkly Rep. 1998; 47(27): 567–70.
- Kajon A.E., Mistchenko A.S., Videla C., Hortal M., Wadell G., Avendaño L.F. Molecular epidemiology of adenovirus acute lower respiratory infections of children in the south cone of South America (1991–1994). J. Med. Virol. 1996; 48(2): 151–6. https://doi.org/10.1002/(sici)1096-9071(199602)48:2%3C151::aid-jmv6%3E3.0.co;2-8
- Salama M., Amitai Z., Nutman A., Gottesman-Yekutieli T., Sherbany H., Drori Y., et al. Outbreak of adenovirus type 55 infection in Israel. J. Clin. Virol. 2016; 78: 31–5. https://doi.org/10.1016/j.jcv.2016.03.002
- Jing S., Zhang J., Cao M., Liu M., Yan Y., Zhao S., et al. Household transmission of human adenovirus type 55 in case of fatal acute respiratory disease. Emerg. Infect. Dis. 2019; 25(9): 1756–8. https://doi.org/10.3201/eid2509.181937
- Burtseva E.I., Panova A.D., Kolobukhina L.V., Ignatjeva A.V., Kirillova E.S., Breslav N.V., et al. Epidemic season 2021–2022: Frequency of co-infection by respiratory viral pathogens. Epidemiologiya i infektsionnye bolezni. 2023; 28(2): 67–77. https://doi.org/10.17816/EID321873 (in Russian)
- Tamura K., Stecher G., Kumar S. MEGA11: Molecular Evolutionary Genetics Analysis Version 11. Mol. Biol. Evol. 2021; 38(7): 3022–7. https://doi.org/10.1093/molbev/msab120
- Houng H.S., Lott L., Gong H., Kuschner R.A., Lynch J.A., Metzgar D. Adenovirus microsatellite reveals dynamics of transmission during a recent epidemic of human adenovirus serotype 14 infection. J. Clin. Microbiol. 2009; 47(7): 2243–8. https://doi.org/10.1128/JCM.01659-08
- Kurskaya O.G., Prokopyeva E.A., Dubovitskiy N.A., Solomatina M.V., Sobolev I.A., Derko A.A., et al. Genetic Diversity of the Human Adenovirus C Isolated from Hospitalized Children in Russia (2019-2022). Viruses. 2024; 16(3): 386. https://doi.org/10.3390/v16030386
- Adenovirus infections, 2008 to 2020, Japan. IASR. 2021; 42(4): 67–9. Available at: https://id-info.jihs.go.jp/niid/en/iasr/12459-494te.html
- Sun H., Hu W., Wei Y., Hao Y. Review: Drawing on the development experiences of infectious disease surveillance systems around the world. China CDC Wkly. 2024; 6(41): 1065–74. https://doi.org/10.46234/ccdcw2024.220
- Ko J.H., Woo H.T., Oh H.S., Moon S.M., Choi J.Y., Lim J.U., et al. Ongoing outbreak of human adenovirus-associated acute respiratory illness in the Republic of Korea military, 2013 to 2018. Korean J. Intern. Med. 2021; 36(1): 205–13. https://doi.org/10.3904/kjim.2019.092
- Hughes J.J., Yang Y., Fries A.C., Maljkovic Berry I., Pollio A.R., Fung C.K., et al. Complete genome sequences of two human adenovirus type 55 isolates from South Korea and the United States. Microbiol. Resour. Announc. 2021; 10(5): e01347-20. https://doi.org/10.1128/MRA.01347-20
- Niang M.N., Diop N.S., Fall A., Kiori D.E., Sarr F.D., Sy S., et al. Respiratory viruses in patients with influenza-like illness in Senegal: Focus on human respiratory adenoviruses. PLoS One. 2017; 12(3): e0174287. https://doi.org/10.1371/journal.pone.0174287
- Hang J., Kajon A.E., Graf P.C.F., Berry I.M., Yang Y., Sanborn M.A., et al. Human adenovirus type 55 distribution, regional persistence, and genetic variability. Emerg. Infect. Dis. 2020; 26(7): 1497–505. https://doi.org/10.3201/eid2607.191707
- Wodrich H., Henaff D., Jammart B., Segura-Morales C., Seelmeir S., Coux O., et al. A capsid-encoded PPxY-motif facilitates adenovirus entry. PLoS Pathog. 2010; 6(3): e1000808. https://doi.org/10.1371/journal.ppat.1000808
- Wang F., De R., Han Z., Xu Y., Zhu R., Sun Y., et al. High-frequency recombination of human adenovirus in children with acute respiratory tract infections in Beijing, China. Viruses. 2024; 16(6): 828. https://doi.org/10.3390/v16060828
补充文件