Генетическая характеристика изолята аденовируса человека 55-го генотипа (Adenoviridae: Mastadenovirus), выделенного в Москве в 2022 г.
- Авторы: Шеин Д.А.1, Рыжова Н.Н.1, Кунда М.С.1, Ермолова Е.И.1, Ожаровская Т.А.1, Попова О.1, Никитенко Н.А.1, Краснослободцев К.Г.1, Бурцева Е.И.1, Зубкова О.В.1, Воронина О.Л.1, Гинцбург А.Л.1
-
Учреждения:
- ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
- Выпуск: Том 70, № 5 (2025)
- Страницы: 431-443
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://journals.rcsi.science/0507-4088/article/view/353626
- DOI: https://doi.org/10.36233/0507-4088-297
- EDN: https://elibrary.ru/aatdbs
- ID: 353626
Цитировать
Аннотация
Введение. Аденовирусная инфекция встречается повсеместно в виде спорадических случаев и отдельных вспышек. Аденовирус человека 55-го генотипа (HAdV-55), эндемичный для территорий Китая и Южной Кореи, вызывает острые респираторные вирусные инфекции (ОРВИ) разной степени тяжести как среди гражданского населения, так и в воинских коллективах в разных странах мира. Геномные исследования способствуют достоверной идентификации HAdV-55.
Цель данной работы состояла в идентификации HAdV, выделенного в Москве в 2022 г., проведении полногеномного секвенирования и сравнительного геномного исследования.
Материалы и методы. HAdV-55 выделили из образца пациента, госпитализированного с пневмонией, исследовали методами анализа полиморфизма длины рестрикционных фрагментов и полногеномного секвенирования. Биоинформационный сравнительный анализ выполняли для выборки геномов 83 изолятов.
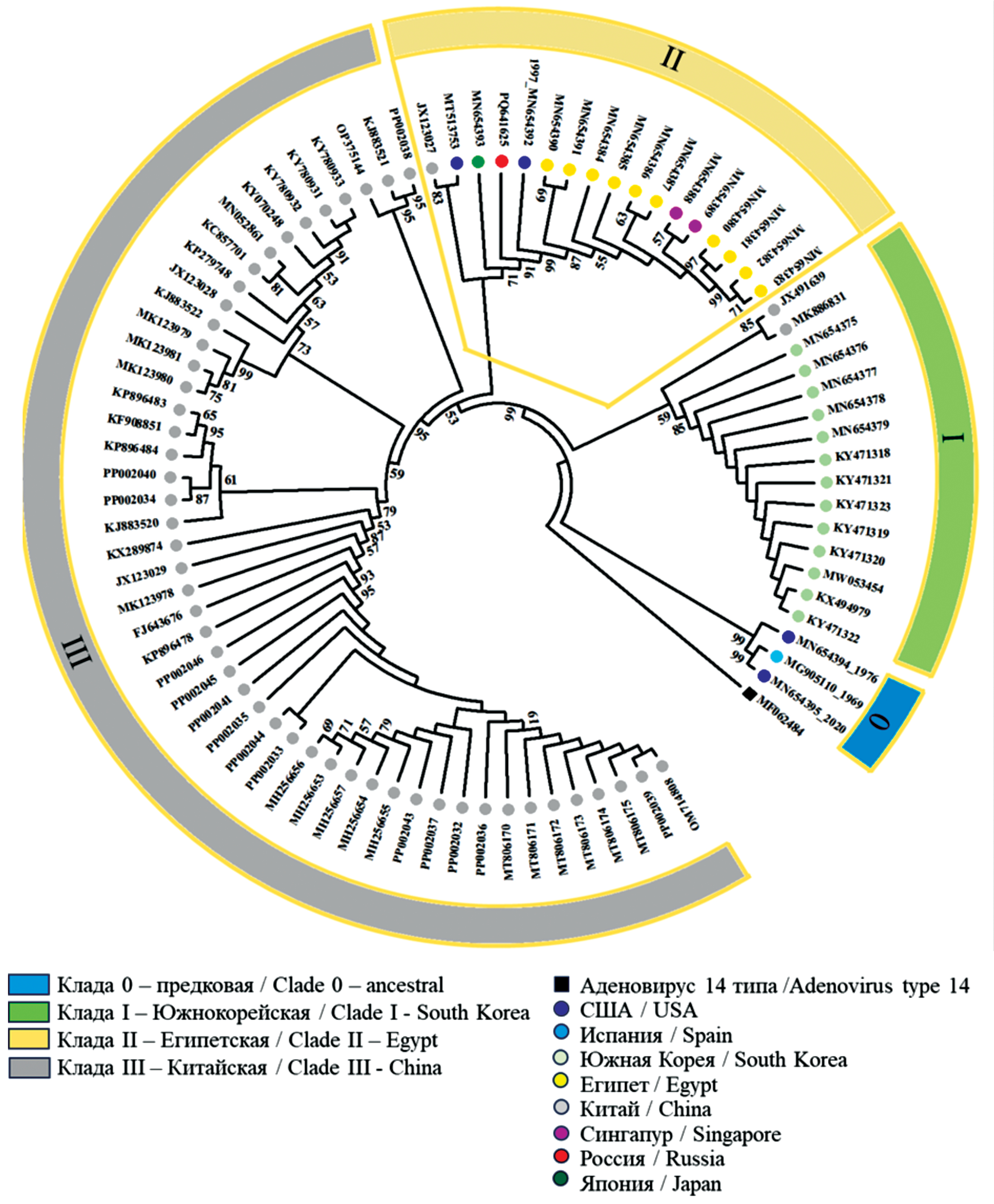
Результаты. Проведено полногеномное секвенирование изолята HAdV-55, впервые выделенного в РФ. Последовательность генома изолята SCV3008:Ad55 депонировали в GenBank (регистрационный номер PQ641625). Выявлены уникальные мутации в геноме SCV3008:Ad55, одна из которых приводила к консервативной замене T29A в пентоне, не влияющей на его функции. Филогенетический анализ показал кластеризацию SCV3008:Ad55 с изолятами клады II, включившей представителей 7 стран разных континентов, что свидетельствует о широком распространении HAdV-55. Изоляты эндемичных регионов Китая и Южной Кореи формировали отдельные клады. Исследование полиморфизма длин микросателлитов в нетранслируемых областях генома стало дополнительным инструментов различения близкородственных геномов.
Заключение. Сравнительное геномное исследование изолятов HAdV-55, появившегося в результате рекомбинации HAdV-14 и HAdV-11, показало медленное накопление мутаций с 1969 г. как в транслируемых, так и в нетранслируемых областях, позволило выявить уникальные замены нового изолята SVC3008:Ad55. Полученная геномная информация заложила основу для дальнейшего мониторинга HAdV-55 в России и продемонстрировала информативность и значимость полногеномных исследований для наблюдения за аденовирусами. Разработка и внедрение в практику методов генотипирования, нацеленных на выявление HAdV-55 и других клинически значимых генотипов, позволит значительно повысить эффективность диагностики аденовирусных инфекций с угрозой развития бронхопневмонии.
Полный текст
Открыть статью на сайте журналаОб авторах
Даниил Алексеевич Шеин
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: daniil.schein@yandex.ru
ORCID iD: 0009-0003-3768-9817
аспирант лаборатории иммунобиотехнологии
Россия, 123098, МоскваНаталья Николаевна Рыжова
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: rynatalia@yandex.ru
ORCID iD: 0000-0001-5361-870X
канд. биол. наук, старший научный сотрудник лаборатории анализа геномов
Россия, 123098, МоскваМарина Сергеевна Кунда
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: markunda99@gmail.com
ORCID iD: 0000-0003-1945-0397
канд. биол. наук, старший научный сотрудник лаборатории анализа геномов
Россия, 123098, МоскваЕкатерина Ивановна Ермолова
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: aksenova16@yandex.ru
ORCID iD: 0000-0002-0437-9404
канд. биол. наук, старший научный сотрудник лаборатории анализа геномов
Россия, 123098, МоскваТатьяна Андреевна Ожаровская
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: t.ozh@yandex.ru
ORCID iD: 0000-0001-7147-1553
канд. биол. наук, старший научный сотрудник лаборатории иммунобиотехнологии
Россия, 123098, МоскваОльга Попова
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: olga.popova31@yandex.ru
ORCID iD: 0000-0003-3248-1227
младший научный сотрудник лаборатории иммунобиотехнологии
Россия, 123098, МоскваНаталья Анатольевна Никитенко
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: nan-nikitenko@yandex.ru
ORCID iD: 0000-0001-5829-744X
канд. биол. наук, старший научный сотрудник лаборатории клеточной микробиологии, заведующая медицинским отделом
Россия, 123098, МоскваКирилл Геннадьевич Краснослободцев
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: kkg_87@mail.ru
ORCID iD: 0000-0003-1745-9128
научный сотрудник этиологии и эпидемиологии гриппа
Россия, 123098, МоскваЕлена Ивановна Бурцева
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: elena-burtseva@yandex.ru
ORCID iD: 0000-0003-2518-6801
д-р мед. наук, заведующая лабораторией этиологии и эпидемиологии гриппа
Россия, 123098, МоскваОльга Вадимовна Зубкова
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Автор, ответственный за переписку.
Email: olga-zubkova@yandex.ru
ORCID iD: 0000-0001-7893-8419
канд. биол. наук, ведущий научный сотрудник лаборатории иммунобиотехнологии
Россия, 123098, МоскваОльга Львовна Воронина
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: olv550@gmail.com
ORCID iD: 0000-0001-7206-3594
канд. биол. наук, доцент, ведущий научный сотрудник – заведующая лабораторией анализа геномов
Россия, 123098, МоскваАлександр Леонидович Гинцбург
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: gintsburg@gamaleya.org
ORCID iD: 0000-0003-1769-5059
д-р биол. наук, академик РАН, профессор, директор
Россия, 123098, МоскваСписок литературы
- Lynch J.P. 3rd, Kajon A.E. Adenovirus: epidemiology, global spread of novel serotypes, and advances in treatment and prevention. Semin. Respir. Crit. Care Med. 2016; 37(4): 586–602. https://doi.org/10.1055/s-0036-1584923
- Coleman K.K., Wong C.C., Jayakumar J., Nguyen T.T., Wong A.W.L., Yadana S., et al. Adenoviral infections in Singapore: Should new antiviral therapies and vaccines be adopted? J. Infect. Dis. 2020; 221(4): 566–77. https://doi.org/10.1093/infdis/jiz489
- Xu W., Xu Z., Huang L., Qin E.Q., Zhang J.L., Zhao P., et al. Transcriptome sequencing identifies novel immune response genes highly related to the severity of human adenovirus type 55 infection. Front. Microbiol. 2019; 10: 130. https://doi.org/10.3389/fmicb.2019.00130
- Kajon A.E., Lamson D.M., St. George K. Emergence and re-emergence of respiratory adenoviruses in the United States. Curr. Opin. Virol. 2019; 34: 63–9. https://doi.org/10.1016/j.coviro.2018.12.004
- Dhingra A., Hage E., Ganzenmueller T., Böttcher S., Hofmann J., Hamprecht K., et al. Molecular Evolution of Human Adenovirus (HAdV) Species C. Sci Rep. 2019; 9(1): 1039. https://doi.org/10.1038/s41598-018-37249-4
- Scott M.K., Chommanard C., Lu X., Appelgate D., Grenz L., Schneider E., et al. Human adenovirus associated with severe respiratory infection, Oregon, USA, 2013–2014. Emerg. Infect. Dis. 2016; 22(6): 1044–51. https://doi.org/10.3201/eid2206.151898
- Hierholzer J.C., Pumarola A., Rodriguez-Torres A., Beltran M. Occurrence of respiratory illness due to an atypical strain of adenovirus type 11 during a large outbreak in Spanish military recruits. Am. J. Epidemiol. 1974; 99(6): 434–42. https://doi.org/10.1093/oxfordjournals.aje.a121632
- Li Q.G., Hambraeus J., Wadell G. Genetic relationship between thirteen genome types of adenovirus 11, 34, and 35 with different tropisms. Intervirology. 1991; 32(6): 338–50. https://doi.org/10.1159/000150218
- Yang Z., Zhu Z., Tang L., Wang L., Tan X., Yu P., et al. Genomic analyses of recombinant adenovirus type 11a in China. J. Clin. Microbiol. 2009; 47(10): 3082–90. https://doi.org/10.1128/JCM.00282-09
- Seto D., Chodosh J., Brister J.R., Jones M.S. Using the whole-genome sequence to characterize and name human adenoviruses. J. Virol. 2011; 85(11): 5701–2. https://doi.org/10.1128/JVI.00354-11
- Seto D., Jones M.S., Dyer D.W., Chodosh J. Characterizing, typing, and naming human adenovirus type 55 in the era of whole genome data. J. Clin. Virol. 2013; 58(4): 741–2. https://doi.org/10.1016/j.jcv.2013.09.025
- Centers for Disease Control and Prevention (CDC). Civilian outbreak of adenovirus acute respiratory disease – South Dakota, 1997. MMWR Morb. Mortal. Wkly Rep. 1998; 47(27): 567–70.
- Kajon A.E., Mistchenko A.S., Videla C., Hortal M., Wadell G., Avendaño L.F. Molecular epidemiology of adenovirus acute lower respiratory infections of children in the south cone of South America (1991–1994). J. Med. Virol. 1996; 48(2): 151–6. https://doi.org/10.1002/(sici)1096-9071(199602)48:2%3C151::aid-jmv6%3E3.0.co;2-8
- Salama M., Amitai Z., Nutman A., Gottesman-Yekutieli T., Sherbany H., Drori Y., et al. Outbreak of adenovirus type 55 infection in Israel. J. Clin. Virol. 2016; 78: 31–5. https://doi.org/10.1016/j.jcv.2016.03.002
- Jing S., Zhang J., Cao M., Liu M., Yan Y., Zhao S., et al. Household transmission of human adenovirus type 55 in case of fatal acute respiratory disease. Emerg. Infect. Dis. 2019; 25(9): 1756–8. https://doi.org/10.3201/eid2509.181937
- Бурцева Е.И., Панова А.Д., Колобухина Л.В., Игнатьева А.В., Кириллова Е.С., Бреслав Н.В. и др. Эпидемический сезон 2021–2022 годов. Частота ко-инфекции респираторными вирусными патогенами. Эпидемиология и инфекционные болезни. 2023; 28(2): 67–77. https://doi.org/10.17816/EID321873
- Tamura K., Stecher G., Kumar S. MEGA11: Molecular Evolutionary Genetics Analysis Version 11. Mol. Biol. Evol. 2021; 38(7): 3022–7. https://doi.org/10.1093/molbev/msab120
- Houng H.S., Lott L., Gong H., Kuschner R.A., Lynch J.A., Metzgar D. Adenovirus microsatellite reveals dynamics of transmission during a recent epidemic of human adenovirus serotype 14 infection. J. Clin. Microbiol. 2009; 47(7): 2243–8. https://doi.org/10.1128/JCM.01659-08
- Kurskaya O.G., Prokopyeva E.A., Dubovitskiy N.A., Solomatina M.V., Sobolev I.A., Derko A.A., et al. Genetic Diversity of the Human Adenovirus C Isolated from Hospitalized Children in Russia (2019-2022). Viruses. 2024; 16(3): 386. https://doi.org/10.3390/v16030386
- Adenovirus infections, 2008 to 2020, Japan. IASR. 2021; 42(4): 67–9. Available at: https://id-info.jihs.go.jp/niid/en/iasr/12459-494te.html
- Sun H., Hu W., Wei Y., Hao Y. Review: Drawing on the development experiences of infectious disease surveillance systems around the world. China CDC Wkly. 2024; 6(41): 1065–74. https://doi.org/10.46234/ccdcw2024.220
- Ko J.H., Woo H.T., Oh H.S., Moon S.M., Choi J.Y., Lim J.U., et al. Ongoing outbreak of human adenovirus-associated acute respiratory illness in the Republic of Korea military, 2013 to 2018. Korean J. Intern. Med. 2021; 36(1): 205–13. https://doi.org/10.3904/kjim.2019.092
- Hughes J.J., Yang Y., Fries A.C., Maljkovic Berry I., Pollio A.R., Fung C.K., et al. Complete genome sequences of two human adenovirus type 55 isolates from South Korea and the United States. Microbiol. Resour. Announc. 2021; 10(5): e01347-20. https://doi.org/10.1128/MRA.01347-20
- Niang M.N., Diop N.S., Fall A., Kiori D.E., Sarr F.D., Sy S., et al. Respiratory viruses in patients with influenza-like illness in Senegal: Focus on human respiratory adenoviruses. PLoS One. 2017; 12(3): e0174287. https://doi.org/10.1371/journal.pone.0174287
- Hang J., Kajon A.E., Graf P.C.F., Berry I.M., Yang Y., Sanborn M.A., et al. Human adenovirus type 55 distribution, regional persistence, and genetic variability. Emerg. Infect. Dis. 2020; 26(7): 1497–505. https://doi.org/10.3201/eid2607.191707
- Wodrich H., Henaff D., Jammart B., Segura-Morales C., Seelmeir S., Coux O., et al. A capsid-encoded PPxY-motif facilitates adenovirus entry. PLoS Pathog. 2010; 6(3): e1000808. https://doi.org/10.1371/journal.ppat.1000808
- Wang F., De R., Han Z., Xu Y., Zhu R., Sun Y., et al. High-frequency recombination of human adenovirus in children with acute respiratory tract infections in Beijing, China. Viruses. 2024; 16(6): 828. https://doi.org/10.3390/v16060828
Дополнительные файлы