Генетическое разнообразие метапневмовируса человека (Pneumoviridae: Metapneumovirus) в России: результаты молекулярного анализа
- Авторы: Фадеев А.В.1, Иванов Я.В.1, Петрова П.А.1, Передерий А.А.1, Писарева М.М.1, Мошкин А.Д.2, Комиссаров А.Б.1, Даниленко Д.М.1, Лиознов Д.А.1
-
Учреждения:
- ФГБУ «НИИ гриппа им. А.А. Смородинцева» Минздрава России
- Научно-исследовательский институт вирусологии ФГБНУ «Федеральный исследовательский центр фундаментальной и трансляционной медицины»
- Выпуск: Том 70, № 2 (2025)
- Страницы: 164-176
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://journals.rcsi.science/0507-4088/article/view/310655
- DOI: https://doi.org/10.36233/0507-4088-294
- EDN: https://elibrary.ru/vfpkwg
- ID: 310655
Цитировать
Аннотация
Актуальность. Метапневмовирус человека (Human metapneumovirus, hMPV) имеет большое эпидемиологическое значение, являясь доминирующей причиной инфекций нижних дыхательных путей у детей младше 2 лет и лиц старше 65 лет. Возможно многократное инфицирование hMPV в течение жизни человека ввиду антигенной и генетической изменчивости возбудителя. При этом генетическая изменчивость hMPV, циркулирующих в России, остается неизученной.
Цель работы. Апробация протокола полногеномного секвенирования hMPV для оценки генетического разнообразия метапневмовирусов, циркулирующих в отдельных субъектах России.
Материалы и методы. Исследовали назофарингеальные мазки от пациентов разного возраста с острыми респираторными вирусными инфекциями, положительные в полимеразной цепной реакции на hMPV. Из части образцов вирус выделяли на клеточной культуре. На платформе Illumina MiSeq проведено полногеномное секвенирование вирусов hMPV из мазков и изолятов с последующим филогенетическим анализом.
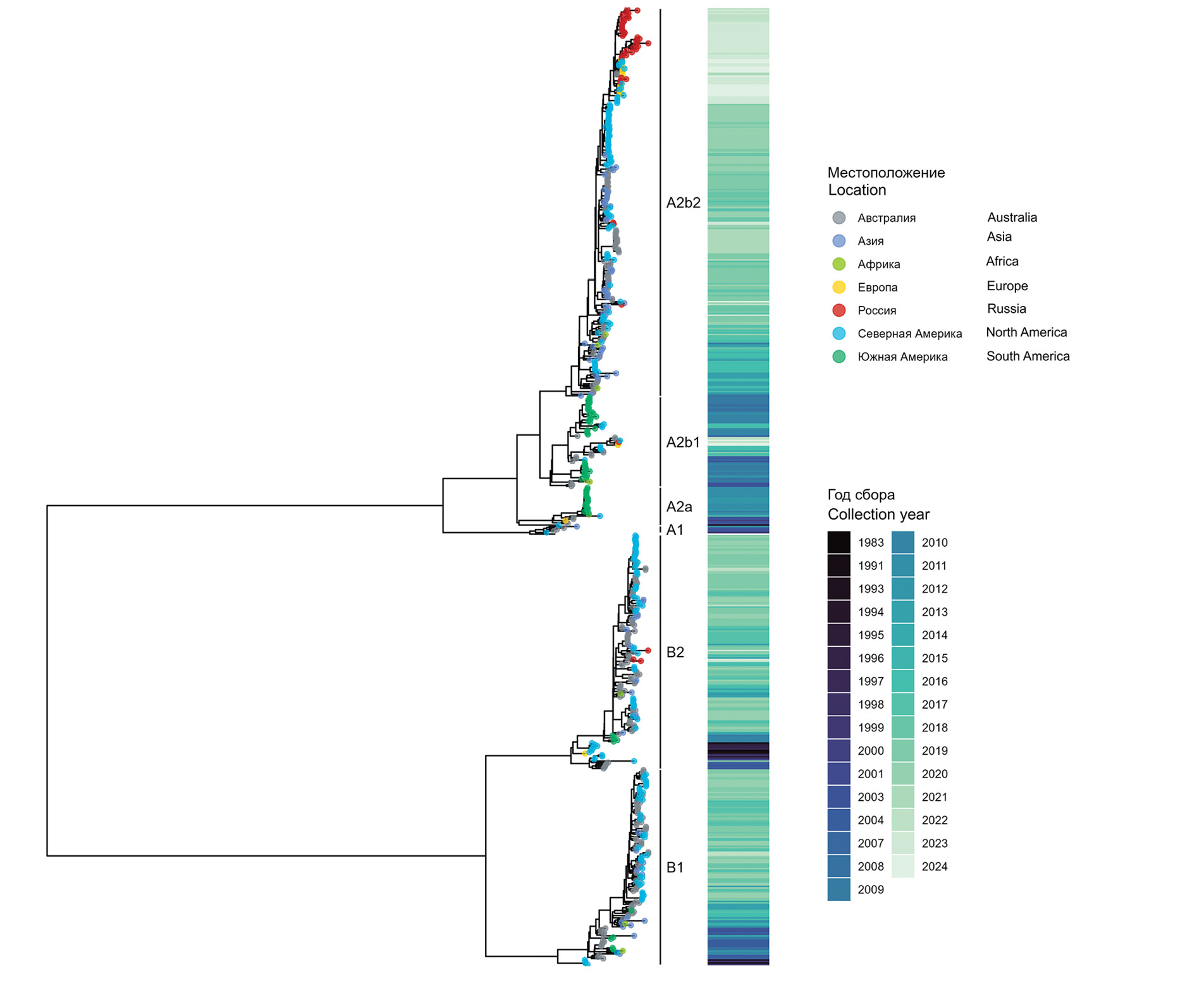
Результаты. Впервые в России проведено полногеномное секвенирование 44 hMPV, циркулировавших в период с 2017 по 2024 г., описана их принадлежность к генетическим группам, показано доминирование клайда A2b2. Подтверждено, что наибольшее разнообразие в генах, кодирующих поверхностные белки вируса, отмечено для гена G, в то время как в гене F в изучаемый период изменения были минимальны.
Заключение. Проведенное исследование дает представление о генетическом разнообразии вирусов hMPV, циркулирующих в отдельных субъектах Российской Федерации. Изучение генетической изменчивости hMPV имеет решающее значение для понимания вирусной эволюции, динамики передачи и механизмов иммунного ускользания, которые влияют на разработку вакцин и противовирусных препаратов.
Ключевые слова
Полный текст
Открыть статью на сайте журналаОб авторах
Артём Викторович Фадеев
ФГБУ «НИИ гриппа им. А.А. Смородинцева» Минздрава России
Email: artem.fadeev@influenza.spb.ru
ORCID iD: 0000-0003-3558-3261
старший научный сотрудник лаборатории молекулярной вирусологии
Россия, 197376, г. Санкт-ПетербургЯн Владимирович Иванов
ФГБУ «НИИ гриппа им. А.А. Смородинцева» Минздрава России
Email: ivanov.yan.vladimirovich@yandex.ru
ORCID iD: 0009-0006-0187-4000
аспирант 2-го года обучения лаборатории молекулярной вирусологии ФГБУ
Россия, 197376, г. Санкт-ПетербургПолина Александровна Петрова
ФГБУ «НИИ гриппа им. А.А. Смородинцева» Минздрава России
Email: suddenkovapolina@gmail.com
ORCID iD: 0000-0001-8527-7946
научный сотрудник лаборатории эволюционной изменчивости вирусов гриппа
Россия, 197376, г. Санкт-ПетербургАлександр Александрович Передерий
ФГБУ «НИИ гриппа им. А.А. Смородинцева» Минздрава России
Email: gilagalex@gmail.com
ORCID iD: 0000-0002-5961-1856
лаборант-исследователь лаборатории молекулярной вирусологии
Россия, 197376, г. Санкт-ПетербургМария Михайловна Писарева
ФГБУ «НИИ гриппа им. А.А. Смородинцева» Минздрава России
Email: maria.pisareva@influenza.spb.ru
ORCID iD: 0000-0002-1499-9957
канд. биол. наук, ведущий научный сотрудник лаборатории молекулярной вирусологии
Россия, 197376, г. Санкт-ПетербургАлексей Дмитриевич Мошкин
Научно-исследовательский институт вирусологии ФГБНУ «Федеральный исследовательский центр фундаментальной и трансляционной медицины»
Email: alex.moshkin727@gmail.com
ORCID iD: 0000-0002-1182-8247
младший научный сотрудник
Россия, 630060, г. НовосибирскАндрей Борисович Комиссаров
ФГБУ «НИИ гриппа им. А.А. Смородинцева» Минздрава России
Автор, ответственный за переписку.
Email: andrey.komissarov@influenza.spb.ru
ORCID iD: 0000-0003-1733-1255
заведующий лабораторией молекулярной вирусологии
Россия, 197376, г. Санкт-ПетербургДарья Михайловна Даниленко
ФГБУ «НИИ гриппа им. А.А. Смородинцева» Минздрава России
Email: daria.danilenko@influenza.spb.ru
ORCID iD: 0000-0001-6174-0836
канд. биол. наук, заместитель директора по научной работе
Россия, 197376, г. Санкт-ПетербургДмитрий Анатольевич Лиознов
ФГБУ «НИИ гриппа им. А.А. Смородинцева» Минздрава России
Email: dmitry.lioznov@influenza.spb.ru
ORCID iD: 0000-0003-3643-7354
д-р мед. наук, профессор, директор
Россия, 197376, г. Санкт-ПетербургСписок литературы
- Uddin S., Thomas M. Human Metapneumovirus. StatPearls. StatPearls Publishing LLC; 2025.
- Sanz-Munoz I., Sanchez-de Prada L., Castrodeza-Sanz J., Eiros J.M. Microbiological and epidemiological features of respiratory syncytial virus. Rev. Esp. Quimioter. 2024; 37(3): 209–20. https://doi.org/10.37201/req/006.2024
- Cifuentes-Munoz N., Branttie J., Slaughter K.B., Dutch R.E. Human metapneumovirus induces formation of inclusion bodies for efficient genome replication and transcription. J. Virol. 2017; 91(24): e01282-17. https://doi.org/10.1128/JVI.01282-17
- Shafagati N., Williams J. Human metapneumovirus – what we know now. F1000Res. 2018; 7: 135. https://doi.org/10.12688/f1000research.12625.1
- Leyrat C., Paesen G.C., Charleston J., Renner M., Grimes J.M. Structural insights into the human metapneumovirus glycoprotein ectodomain. J. Virol. 2014; 88(19): 11611–6. https://doi.org/10.1128/JVI.01726-14
- Jesse S.T., Ludlow M., Osterhaus A.D.M.E. Zoonotic origins of human metapneumovirus: a journey from birds to humans. Viruses. 2022; 14(4): 677. https://doi.org/10.3390/v14040677
- Chang A., Masante C., Buchholz U.J., Dutch R.E. Human metapneumovirus (HMPV) binding and infection are mediated by interactions between the HMPV fusion protein and heparan sulfate. J. Virol. 2012; 86(6): 3230–43. https://doi.org/10.1128/JVI.06706-11
- Ribo-Molina P., van Nieuwkoop S., Mykytyn A.Z., van Run P., Lamers M.M., Haagmans B.L., et al. Human metapneumovirus infection of organoid-derived human bronchial epithelium represents cell tropism and cytopathology as observed in in vivo models. mSphere. 2024; 9(2): e0074323. https://doi.org/10.1128/msphere.00743-23
- Kinder J.T., Moncman C.L., Barrett C., Jin H., Kallewaard N., Dutch R.E. Respiratory syncytial virus and human metapneumovirus infections in three-dimensional human airway tissues expose an interesting dichotomy in viral replication, spread, and inhibition by neutralizing antibodies. J. Virol. 2020; 94(20): e01068-20. https://doi.org/10.1128/JVI.01068-20
- Herfst S., de Graaf M., Schickli J.H., Tang R.S., Kaur J., Yang C.F., et al. Recovery of human metapneumovirus genetic lineages a and B from cloned cDNA. J. Virol. 2004; 78(15): 8264–70. https://doi.org/10.1128/JVI.78.15.8264-8270.2004
- Groen K., van Nieuwkoop S., Meijer A., van der Veer B., van Kampen J.J.A., Fraaij P.L., et al. Emergence and Potential extinction of genetic lineages of human metapneumovirus between 2005 and 2021. mBio. 2023; 14(1): e0228022. https://doi.org/10.1128/mbio.02280-22
- Kahn J.S. Epidemiology of human metapneumovirus. Clin. Microbiol. Rev. 2006; 19(3): 546–57. https://doi.org/10.1128/CMR.00014-06
- van den Hoogen B.G., Herfst S., Sprong L., Cane P.A., Forleo-Neto E., de Swart R.L., et al. Antigenic and genetic variability of human metapneumoviruses. Emerg. Infect. Dis. 2004; 10(4): 658–66. https://doi.org/10.3201/eid1004.030393
- Yang C.F., Wang C.K., Tollefson S.J., Piyaratna R., Lintao L.D., Chu M., et al. Genetic diversity and evolution of human metapneumovirus fusion protein over twenty years. Virol. J. 2009; 6: 138. https://doi.org/10.1186/1743-422X-6-138
- Yang C.F., Wang C.K., Tollefson S.J., Lintao L.D., Liem A., Chu M., et al. Human metapneumovirus G protein is highly conserved within but not between genetic lineages. Arch. Virol. 2013; 158(6): 1245–52. https://doi.org/10.1007/s00705-013-1622-x
- van den Hoogen B.G., de Jong J.C., Groen J., Kuiken T., de Groot R., Fouchier R.A., et al. A newly discovered human pneumovirus isolated from young children with respiratory tract disease. Nat. Med. 2001; 7(6): 719–24. https://doi.org/10.1038/89098
- Feng Y., He T., Zhang B., Yuan H., Zhou Y. Epidemiology and diagnosis technologies of human metapneumovirus in China: a mini review. Virol. J. 2024; 21(1): 59. https://doi.org/10.1186/s12985-024-02327-9
- Esposito S., Mastrolia M.V. Metapneumovirus infections and respiratory complications. Semin. Respir. Crit. Care Med. 2016; 37(4): 512–21. https://doi.org/10.1055/s-0036-1584800
- Hamelin M.E., Abed Y., Boivin G. Human metapneumovirus: a new player among respiratory viruses. Clin. Infect. Dis. 2004; 38(7): 983–90. https://doi.org/10.1086/382536
- Yi L., Zou L., Peng J., Yu J., Song Y., Liang L., et al. Epidemiology, evolution and transmission of human metapneumovirus in Guangzhou China, 2013–2017. Sci. Rep. 2019; 9(1): 14022. https://doi.org/10.1038/s41598-019-50340-8
- Matsuzaki Y., Itagaki T., Ikeda T., Aoki Y., Abiko C., Mizuta K. Human metapneumovirus infection among family members. Epidemiol. Infect. 2013; 141(4): 827–32. https://doi.org/10.1017/S095026881200129X
- Hacker K., Kuan G., Vydiswaran N., Chowell-Puente G., Patel M., Sanchez N., et al. Pediatric burden and seasonality of human metapneumovirus over 5 years in Managua, Nicaragua. Influenza Other Respir. Viruses. 2022; 16(6): 1112–21. https://doi.org/10.1111/irv.13034
- Howard L.M., Edwards K.M., Zhu Y., Griffin M.R., Weinberg G.A., Szilagyi P.G., et al. Clinical features of human metapneumovirus infection in ambulatory children aged 5-13 years. J. Pediatric Infect. Dis. Soc. 2018; 7(2): 165–8. https://doi.org/10.1093/jpids/pix012
- Шарипова Е.В., Бабаченко И.В., Орлова Е.Д. Метапневмовирусная инфекция у детей. Педиатр. 2020; 11(5): 13–9. https://doi.org/10.17816/PED11513-19 https://elibrary.ru/xqnxgf
- Lu G., Gonzalez R., Guo L., Wu C., Wu J., Vernet G., et al. Large-scale seroprevalence analysis of human metapneumovirus and human respiratory syncytial virus infections in Beijing, China. Virol. J. 2011; 8: 62. https://doi.org/10.1186/1743-422X-8-62
- Яцышина СБ. Пневмовирусы в инфекционной патологии человека. Журнал микробиологии, эпидемиологии и иммунобиологии. 2017; 94(6): 95–105. https://doi.org/10.36233/0372-9311-2017-6-95-105 https://elibrary.ru/zaddtv
- Sugimoto S., Kawase M., Suwa R., Kakizaki M., Kume Y., Chishiki M., et al. Development of a duplex real-time RT-PCR assay for the detection and identification of two subgroups of human metapneumovirus in a single tube. J. Virol. Methods. 2023; 322: 114812. https://doi.org/10.1016/j.jviromet.2023.114812
- Groen K., van Nieuwkoop S., Bestebroer T.M., Fraaij P.L., Fouchier R.A.M., van den Hoogen B.G. Whole genome sequencing of human metapneumoviruses from clinical specimens using MinION nanopore technology. Virus Res. 2021; 302: 198490. https://doi.org/10.1016/j.virusres.2021.198490
- Aksamentov I., Roemer C., Hodcroft E., Neher R. Nextclade: clade assignment, mutation calling and quality control for viral genomes. J. Open Source Softw. 2021; 6(67): 3773. https://doi.org/10.21105/joss.03773
- Rambaut A., Lam T.T., Max Carvalho L., Pybus O.G. Exploring the temporal structure of heterochronous sequences using TempEst (formerly Path-O-Gen). Virus Evol. 2016; 2(1): vew007. https://doi.org/10.1093/ve/vew007
- McConnell B.S., Parker M.W. Protein intrinsically disordered regions have a non-random, modular architecture. Bioinformatics. 2023; 39(12): btad732. https://doi.org/10.1093/bioinformatics/btad732
- Parida P., Sudheesh N., Sanjay E.R., Jagadesh A., Marate S., Govindakaranavar A. The emergence of human metapneumovirus G gene duplication in hospitalized patients with respiratory tract infection, India, 2016–2018. Mol. Biol. Rep. 2023; 50(2): 1109–16. https://doi.org/10.1007/s11033-022-08092-8
- Piñana M., Vila J., Maldonado C., Galano-Frutos J.J., Valls M., Sancho J., et al. Insights into immune evasion of human metapneumovirus: novel 180- and 111-nucleotide duplications within viral G gene throughout 2014–2017 seasons in Barcelona, Spain. J. Clin. Virol. 2020; 132: 104590. https://doi.org/10.1016/j.jcv.2020.104590
- Jagusic M., Slovic A., Ivancic-Jelecki J., Ljubin-Sternak S., Vilibić-Čavlek T., Tabain I., et al. Molecular epidemiology of human respiratory syncytial virus and human metapneumovirus in hospitalized children with acute respiratory infections in Croatia, 2014–2017. Infect. Genet. Evol. 2019; 76: 104039. https://doi.org/10.1016/j.meegid.2019.104039
- Saikusa M., Kawakami C., Nao N., Takeda M., Usuku S., Sasao T., et al. 180-nucleotide duplication in the G gene of human metapneumovirus A2b subgroup strains circulating in Yokohama city, Japan, since 2014. Front. Microbiol. 2017; 8: 402. https://doi.org/10.3389/fmicb.2017.00402
- Saikusa M., Nao N., Kawakami C., Usuku S., Sasao T., Toyozawa T., et al. A novel 111-nucleotide duplication in the G gene of human metapneumovirus. Microbiol. Immunol. 2017; 61(11): 507–12. https://doi.org/10.1111/1348-0421.12543
- Yi L., Zou L., Peng J., Yu J., Song Y., Liang L., et al. Epidemiology, evolution and transmission of human metapneumovirus in Guangzhou China, 2013–2017. Sci. Rep. 2019; 9(1): 14022. https://doi.org/10.1038/s41598-019-50340-8
Дополнительные файлы