Phylogenetic analysis of variants of the Puumala virus (Hantaviridae: Orthohantavirus) circulating in the Saratov region
- Authors: Krasnov Y.M.1, Naidenova E.V.1, Guseva N.P.1, Polunina T.A.1, Sharapova N.A.1, Sosedova E.A.1, Kotova N.V.1, Zakharov K.S.1, Kazantsev A.V.1, Domanina I.V.1, Chekashov V.N.1, Shilov M.M.1, Kondratiev E.N.1, Osina N.A.1, Kutyrev V.V.1
-
Affiliations:
- Russian Research Anti-Plague Institute «Microbe»
- Issue: Vol 69, No 2 (2024)
- Pages: 162-174
- Section: ORIGINAL RESEARCHES
- URL: https://journals.rcsi.science/0507-4088/article/view/256877
- DOI: https://doi.org/10.36233/0507-4088-224
- EDN: https://elibrary.ru/ijmubu
- ID: 256877
Cite item
Abstract
The objective is to determine the complete nucleotide sequence and conduct a phylogenetic analysis of genome variants of the Puumala virus isolated in the Saratov region.
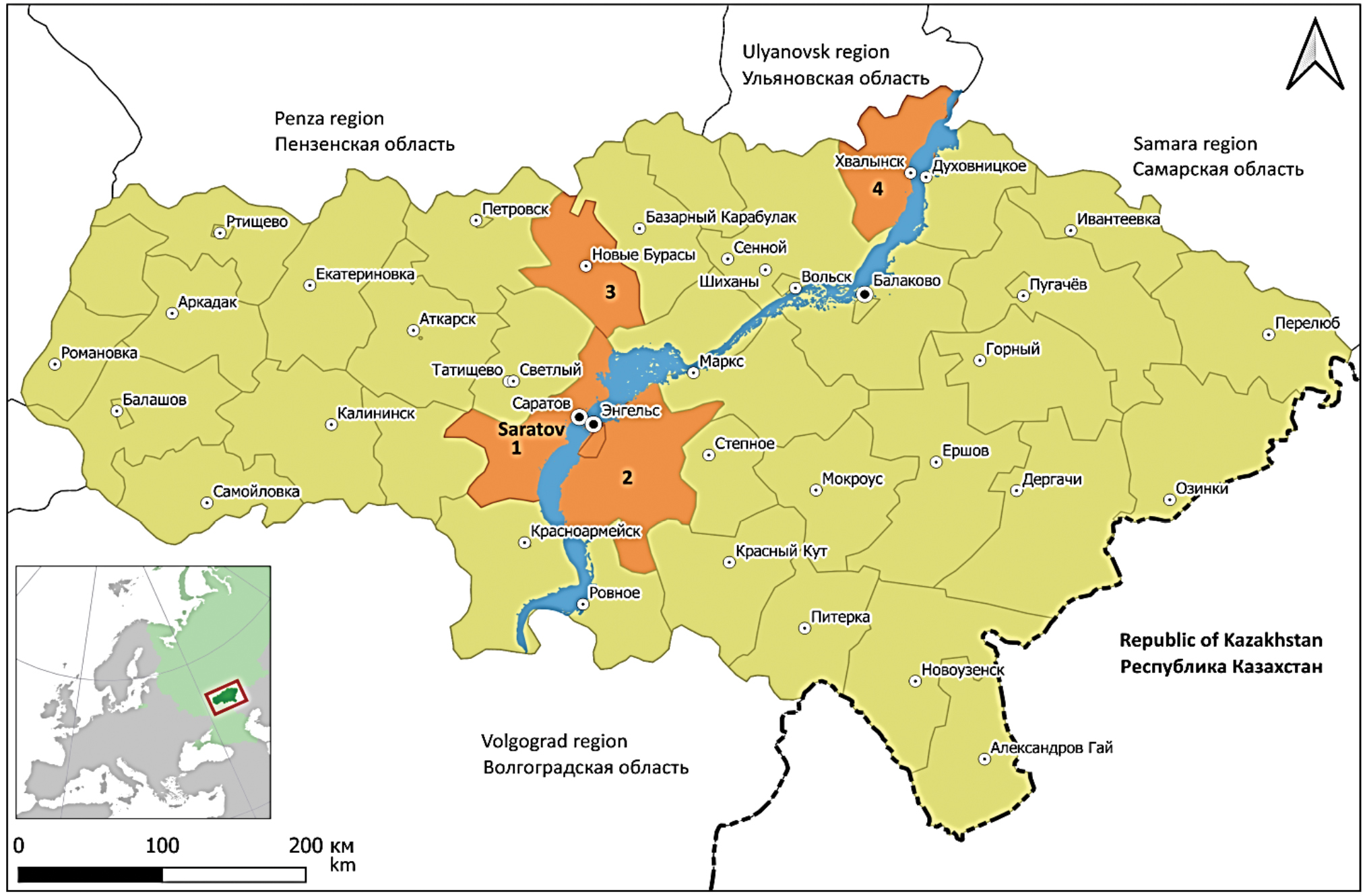
Materials and methods. The samples for the study were field material collected in the Gagarinsky (formerly Saratovsky), Engelssky, Novoburassky and Khvalynsky districts of the Saratov region in the period from 2019 to 2022. To specifically enrich the Puumala virus genome in the samples, were used PCR and developed a specific primer panel. Next, the resulting PCR products were sequenced and the fragments were assembled into one sequence for each segment of the virus genome. To construct phylogenetic trees, the maximum parsimony algorithm was used.
Results. Genetic variants of the Puumala virus isolated in the Saratov region have a high degree of genome similarity to each other, which indicates their unity of origin. According to phylogenetic analysis, they all form a separate branch in the cluster formed by hantaviruses from other subjects of the Volga Federal District. The virus variants from the Republics of Udmurtia and Tatarstan, as well as from the Samara and Ulyanovsk regions, are closest to the samples from the Saratov region.
Conclusion. The data obtained show the presence of a pronounced territorial confinement of strains to certain regions or areas that are the natural biotopes of their carriers. This makes it possible to fairly accurately determine the territory of possible infection of patients and/or the circulation of carriers of these virus variants based on the sequence of individual segments of their genome.
Full Text
##article.viewOnOriginalSite##About the authors
Yaroslav M. Krasnov
Russian Research Anti-Plague Institute «Microbe»
Email: rusrapi@microbe.ru
ORCID iD: 0000-0002-4909-2394
PhD in Chemistry, leading researcher
Russian Federation, 410005, SaratovEkaterina V. Naidenova
Russian Research Anti-Plague Institute «Microbe»
Author for correspondence.
Email: katim2003@mail.ru
ORCID iD: 0000-0001-6474-3696
PhD in Biology, leading researcher
Russian Federation, 410005, SaratovNatalia P. Guseva
Russian Research Anti-Plague Institute «Microbe»
Email: rusrapi@microbe.ru
ORCID iD: 0000-0003-3763-9708
PhD in Biology, senior researcher
Russian Federation, 410005, SaratovTatyana A. Polunina
Russian Research Anti-Plague Institute «Microbe»
Email: rusrapi@mail.ru
ORCID iD: 0000-0002-2234-2760
PhD in Medical sciences, senior researcher
Russian Federation, 410005, SaratovNatalya A. Sharapova
Russian Research Anti-Plague Institute «Microbe»
Email: rusrapi@microbe.ru
ORCID iD: 0000-0002-5289-7783
PhD in Biology, researcher
Russian Federation, 410005, SaratovEkaterina A. Sosedova
Russian Research Anti-Plague Institute «Microbe»
Email: rusrapi@microbe.ru
ORCID iD: 0009-0004-4443-2646
Researcher
Russian Federation, 410005, SaratovNina V. Kotova
Russian Research Anti-Plague Institute «Microbe»
Email: rusrapi@microbe.ru
ORCID iD: 0000-0002-9270-523X
Researcher
Russian Federation, 410005, SaratovKirill S. Zakharov
Russian Research Anti-Plague Institute «Microbe»
Email: zaharov_ks@mail.ru
ORCID iD: 0000-0002-4726-309X
PhD in Biology, senior researcher
Russian Federation, 410005, SaratovAndrey V. Kazantsev
Russian Research Anti-Plague Institute «Microbe»
Email: andreikazancev@mail.ru
ORCID iD: 0000-0003-1790-0411
researcher
Russian Federation, 410005, SaratovIrina V. Domanina
Russian Research Anti-Plague Institute «Microbe»
Email: rusrapi@microbe.ru
ORCID iD: 0000-0002-4731-8089
researcher
Russian Federation, 410005, SaratovVladimir N. Chekashov
Russian Research Anti-Plague Institute «Microbe»
Email: rusrapi@microbe.ru
ORCID iD: 0000-0002-9593-4353
PhD in Biology, senior researcher
Russian Federation, 410005, SaratovMikhail M. Shilov
Russian Research Anti-Plague Institute «Microbe»
Email: rusrapi@microbe.ru
ORCID iD: 0000-0002-0083-8212
PhD in Biology, researcher
Russian Federation, 410005, SaratovEvgeniy N. Kondratiev
Russian Research Anti-Plague Institute «Microbe»
Email: rusrapi@microbe.ru
ORCID iD: 0000-0002-7508-4355
Researcher
Russian Federation, 410005, SaratovNatalya A. Osina
Russian Research Anti-Plague Institute «Microbe»
Email: davidova_n_work@mail.ru
ORCID iD: 0000-0003-0954-5683
PhD in Biology, Head of Department of Microbiology
Russian Federation, 410005, SaratovVladimir V. Kutyrev
Russian Research Anti-Plague Institute «Microbe»
Email: rusrapi@mail.ru
ORCID iD: 0000-0003-3788-3452
Academician of RAS, Professor, Director
Russian Federation, 410005, SaratovReferences
- Savitskaya T.A., Ivanova A.V., Isaeva G.Sh., Reshetnikova I.D., Trifonov V.A., Ziatdinov V.B., et al. Analysis of the epidemiological situation of hemorrhagic fever with renal syndrome in the Russian Federation in 2022 and forecast of its development for 2023. Problemy osobo opasnykh infektsii. 2023; (1): 85–95. https://doi.org/10.21055/0370-1069-2023-1-85-95 https://elibrary.ru/mgxnza (in Russian)
- Ivanova A.V., Safronov V.A., Popov N.V., Kuklev E.V. Epidemiological zoning of the Volga federal district territory by the level of potential epidemic hazard of hemorrhagic fever with renal syndrome natural foci. Problemy osobo opasnykh infektsii. 2020; (1): 91–6. https://doi.org/10.21055/0370-1069-2020-1-91-96 https://elibrary.ru/jrywjk (in Russian)
- Chumachkova E.A., Ivanova A.V., Porshakov A.M., Vyatkin I.N., Forostyanaya M.V., Chumachkov K.Ya., et al. Zoning of the territory of the Saratov region by the intensity of epidemic manifestations of HFRS using GIS analysis. Problemy osobo opasnykh infektsii. 2023; (3): 156–63. https://doi.org/10.21055/0370-1069-2023-3-156-163 https://elibrary.ru/dwrlpq (in Russian)
- Tkachenko E.A., Ishmukhametov A.A., Dzagurova T.K., Bernshtein A.D., Morozov V.G., Siniugina A.A., et al. Hemorrhagic fever with renal syndrome, Russia. Emerg. Infect. Dis. 2019; 25(12): 2325–8. https://doi.org/10.3201/eid2512.181649
- Ishmukhametov A.A., Dzagurova T.K., Morozov V.G., Kurashova S.S., Balovneva M.V., Sotskova S.E., et al. Characteristics of hantaviruses as causative agents of the zoonotic hemorrhagic fevers. Epidemiologiya i vaktsinoprofilaktika. 2017; 16(3): 26–32. https://doi.org/10.31631/2073-3046-2017-16-3-26-32 https://elibrary.ru/yrhmch (in Russian)
- ICTV. Taxonomy Browser. Available at: https://ictv.global/taxonomy
- Kabwe E., Davidyuk Y., Shamsutdinov A., Garanina E., Martynova E., Kitaeva K., et al. Orthohantaviruses, emerging zoonotic pathogens. Pathogens. 2020; 9(9): 775. https://doi.org/10.3390/pathogens9090775
- Yashina L.N., Tregubchak T.V., Malyshev B.S., Smetannikova N.A., Grishchenko I.V., Dol’skii A.A., et al. Hantavirus associated with hemorrhagic fever with renal syndrome outbreak in the Saratov region in 2019. Problemy osobo opasnykh infektsii. 2021; (4): 150–6. https://doi.org/10.21055/0370-1069-2021-4-150-156 https://elibrary.ru/dxxsey (in Russian)
- Davidyuk Y.N., Kabwe E., Shamsutdinov A.F., Knyazeva A.V., Martynova E.V., Ismagilova R.K., et al. The Distribution of Puumala orthohantavirus genome variants correlates with the regional landscapes in the Trans-Kama area of the Republic of Tatarstan. Pathogens. 2021; 10(9): 1169. https://doi.org/10.3390/pathogens10091169
- Kabwe E., Shamsutdinov A.F., Suleimanova S., Martynova E.V., Ismagilova R.K., Shakirova V.G., et al. Puumala orthohantavirus reassortant genome variants likely emerging in the watershed forests. Int. J. Mol. Sci. 2023; 24(2): 1018. https://doi.org/10.3390/ijms24021018
- Kabwe E., Al Sheikh W., Shamsutdinov A.F., Ismagilova R.K., Martynova E.V., Ohlopkova O.V., et al. Analysis of Puumala orthohantavirus genome variants identified in the territories of Volga Federal District. Trop. Med. Infect. Dis. 2022; 7(3): 46. https://doi.org/10.3390/tropicalmed7030046
- Blinova E., Deviatkin A., Makenov M., Popova Y., Dzagurova T. Evolutionary formation and distribution of Puumala virus genome variants, Russia. Emerg. Infect. Dis. 2023; 29(7): 1420–4. https://doi.org/10.3201/eid2907.221731
- Castel G., Chevenet F., Razzauti M., Murri S., Marianneau P., Cosson J.F., et al. Phylogeography of Puumala orthohantavirus in Europe. Viruses. 2019; 11(8): 679. https://doi.org/10.3390/v11080679
- Souza W.M., Bello G., Amarilla A.A., Alfonso H.L., Aquino V.H., Figueiredo L.T. Phylogeography and evolutionary history of rodent-borne hantaviruses. Infect. Genet. Evol. 2014; 21: 198–204. https://doi.org/10.1016/j.meegid.2013.11.015
- Ramsden C., Holmes E.C., Charleston M.A. Hantavirus evolution in relation to its rodent and insectivore hosts: no evidence for codivergence. Mol. Biol. Evol. 2009; 26(1): 143–53. https://doi.org/10.1093/molbev/msn234
Supplementary files