Identification by enzyme immunoassay of escape mutants S143L and G145R of hepatitis B virus (Hepadnaviridae: Orthohepadnavirus: Hepatitis B virus)
- Authors: Konopleva M.V.1, Feldsherova A.A.1, Elgort D.A.1, Tupoleva T.A.2, Kokhanovskaya N.A.1, Pankratova V.N.1, Semenenko T.A.1, Suslov A.P.1
-
Affiliations:
- FSBI «National Research Centre for Epidemiology and Microbiology named after honorary academician N.F. Gamaleya» of the Ministry of Health of Russia
- FSBI «National Medical Research Center for Hematology» of the Ministry of Health of Russia
- Issue: Vol 67, No 1 (2022)
- Pages: 48-58
- Section: ORIGINAL RESEARCHES
- URL: https://journals.rcsi.science/0507-4088/article/view/118198
- DOI: https://doi.org/10.36233/0507-4088-91
- ID: 118198
Cite item
Abstract
Introduction. The achievement of the goal of the World Health Organization to eliminate viral hepatitis B by 2030 seems to be problematic partly due to the presence of escape mutants of its etiological agent, hepatitis B virus (HBV) (Hepadnaviridae: Orthohepadnavirus: Hepatitis B virus), that are spreading mainly in the risk groups. Specific routine diagnostic assays aimed at identification of HBV escape mutants do not exist.
The study aimed the evaluation of the serological fingerprinting method adapted for routine detection of escape mutations in 143 and 145 aa positions of HBV surface antigen (HBsAg).
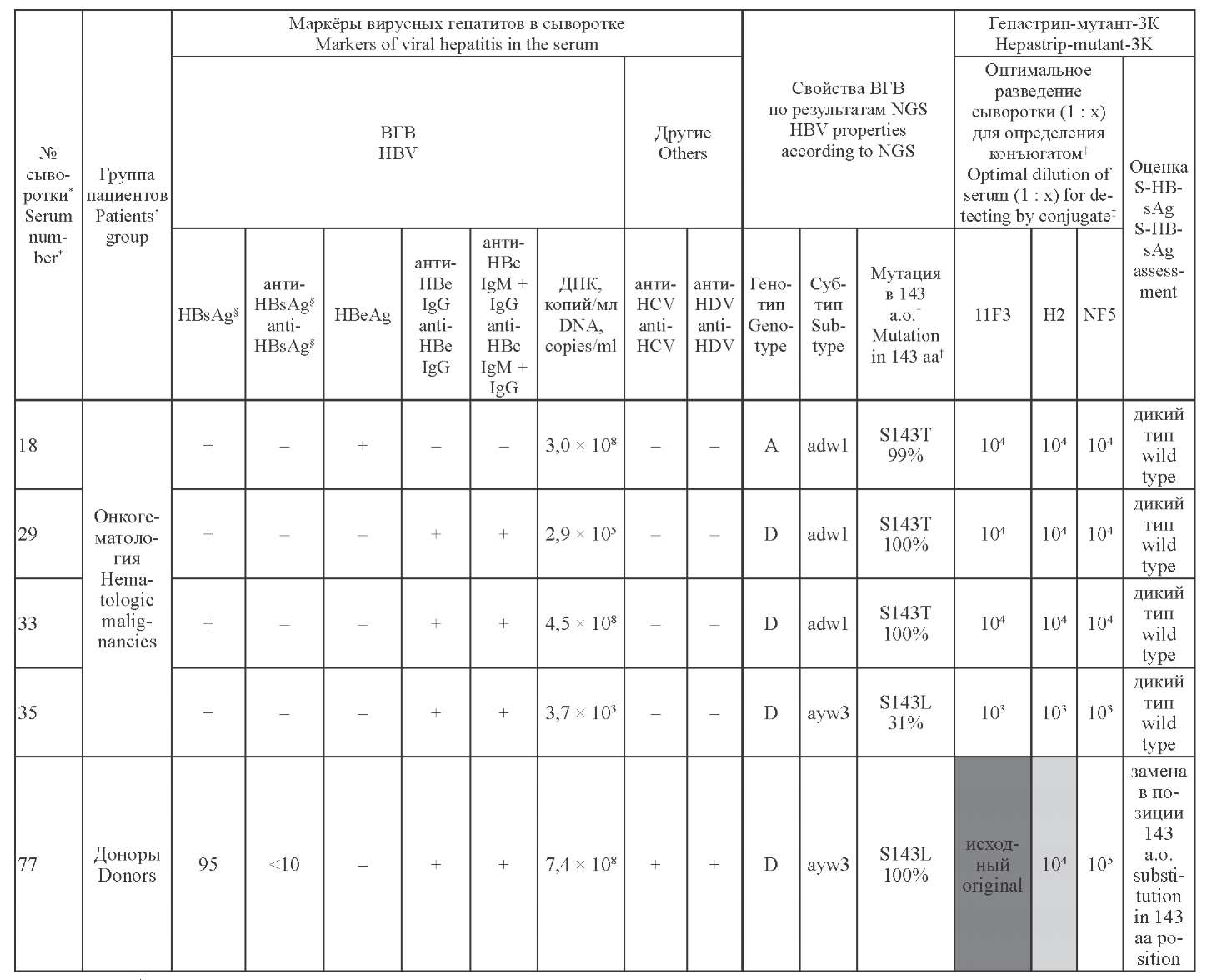
Material and methods. HBV DNA from 56 samples of HBsAg-positive blood sera obtained from donors, chronic HBsAg carriers and oncohematology patients has been sequenced. After the identification of mutations in HBsAg, the samples were tested in the enzyme-linked immunosorbent assay (ELISA) kit «Hepastrip-mutant-3K».
Results and discussion. Escape mutations were detected mainly in patients with hematologic malignancies. Substitutions in 143 and 145 aa were found in 10.81% and in 8.11% of such patients, respectively. The G145R mutation was recognized using ELISA kit in almost all cases. The kit specifically recognized the S143L substitution in contrast to the S143T variant. The presence of neighbor mutation D144E can be assumed due to it special serological fingerprint.
Conclusion. ELISA-based detection of escape mutations S143L, D144E and G145R can be used for routine diagnostics, especially in the risk groups. The diagnostic parameters of the kit can be refined in additional studies. This immunoassay and methodology are applicable for the development and quality control of vaccines against escape mutants.
Full Text
##article.viewOnOriginalSite##About the authors
M. V. Konopleva
FSBI «National Research Centre for Epidemiology and Microbiology named after honorary academician N.F. Gamaleya» of the Ministry of Health of Russia
Author for correspondence.
Email: maria-konopleva@rambler.ru
ORCID iD: 0000-0002-9724-695X
Maria V. Konopleva, Ph.D (Biol.), Senior Researcher of the Immunity Mediators and Effectors Laboratory, Immunology Department
123098, Moscow
Russian FederationA. A. Feldsherova
FSBI «National Research Centre for Epidemiology and Microbiology named after honorary academician N.F. Gamaleya» of the Ministry of Health of Russia
ORCID iD: 0000-0001-7216-4301
123098, Moscow
Russian FederationD. A. Elgort
FSBI «National Research Centre for Epidemiology and Microbiology named after honorary academician N.F. Gamaleya» of the Ministry of Health of Russia
ORCID iD: 0000-0002-2197-4184
123098, Moscow
Russian FederationT. A. Tupoleva
FSBI «National Medical Research Center for Hematology» of the Ministry of Health of Russia
ORCID iD: 0000-0003-4668-9379
125167, Moscow
Russian FederationN. A. Kokhanovskaya
FSBI «National Research Centre for Epidemiology and Microbiology named after honorary academician N.F. Gamaleya» of the Ministry of Health of Russia
ORCID iD: 0000-0001-5142-846X
123098, Moscow
Russian FederationV. N. Pankratova
FSBI «National Research Centre for Epidemiology and Microbiology named after honorary academician N.F. Gamaleya» of the Ministry of Health of Russia
ORCID iD: 0000-0003-4427-1809
123098, Moscow
Russian FederationT. A. Semenenko
FSBI «National Research Centre for Epidemiology and Microbiology named after honorary academician N.F. Gamaleya» of the Ministry of Health of Russia
ORCID iD: 0000-0002-6686-9011
123098, Moscow
Russian FederationA. P. Suslov
FSBI «National Research Centre for Epidemiology and Microbiology named after honorary academician N.F. Gamaleya» of the Ministry of Health of Russia
ORCID iD: 0000-0001-5731-3284
123098, Moscow
Russian FederationReferences
- Mahamat G., Kenmoe S., Akazong E.W., Ebogo-Belobo J.T., Mbaga D.S., Bowo-Ngandji A., et al. Global prevalence of hepatitis B virus serological markers among healthcare workers: A systematic review and meta-analysis. World J. Hepatol. 2021; 13(9): 1190–202. https://doi.org/10.4254/wjh.v13.i9.1190
- Polaris Observatory Collaborators. The case for simplifying and using absolute targets for viral hepatitis elimination goals. J. Viral. Hepat. 2021; 28(1): 12–9. https://doi.org/10.1111/jvh.13412
- Han Q., Zhang C., Zhang J., Tian Z. The role of innate immunity in HBV infection. Semin. Immunopathol. 2013; 35(1): 23–38. https://doi.org/10.1007/s00281-012-0331-y
- Araujo N.M., Teles S.A., Spitz N. Comprehensive Analysis of Clinically Significant Hepatitis B Virus Mutations in Relation to Genotype, Subgenotype and Geographic Region. Front. Microbiol. 2020; 11: 616023. https://doi.org/10.3389/fmicb.2020.616023
- Баженов А.И., Эльгорт Д.А., Фельдшерова А.А., Будницкая П.З., Никитина Г.И., Хац Ю.С., и др. Выявление антител к мутантным формам HBsAg у лиц, иммунизированных против гепатита В вакцинами разных субтипов. Эпидемиология и Вакцинопрофилактика. 2011; 5 (60): 49–53.
- Hossain M.G., Ueda K. A meta-analysis on genetic variability of RT/HBsAg overlapping region of hepatitis B virus (HBV) isolates of Bangladesh. Infect. Agent. Cancer. 2019; 14: 33. https://doi.org/10.1186/s13027-019-0253-6
- Konopleva M.V., Belenikin M.S., Shanko A.V., Bazhenov A.I., Kiryanov S.A., Tupoleva T.A., et al. Detection of S-HBsAg Mutations in Patients with Hematologic Malignancies. Diagnostics (Basel). 2021; 11(6): 969. https://doi.org/10.3390/diagnostics11060969
- Семененко Т.А., Ярош Л.В., Баженов А.И., Никитина Г.Ю., Клейменов Д.А., Эльгорт Д.А., и др. Эпидемиологическая оценка распространенности «скрытых» форм и HBsAg-мутантов вируса гепатита В у гематологических больных. Эпидемиология и Вакцинопрофилактика. 2012; 6 (67): 9–14.
- Hsu H.Y., Chang M.H., Ni Y.H., Chen H.L. Survey of hepatitis B surface variant infection in children 15 years after a nationwide vaccination programme in Taiwan. Gut. 2004; 53(10): 1499–503. https://doi.org/10.1136/gut.2003.034223
- Komatsu H., Inui A., Umetsu S., Tsunoda T., Sogo T., Konishi Y., et al. Evaluation of the G145R Mutant of the Hepatitis B Virus as a Minor Strain in Mother-to-Child Transmission. PLoS One. 2016; 11(11): e0165674. https://doi.org/10.1371/journal.pone.0165674
- Weber B. Genetic variability of the S gene of hepatitis B virus: clinical and diagnostic impact. J. Clin. Virol. 2005, 32(2): 102–12. https://doi.org/10.1016/j.jcv.2004.10.008
- Deguchi M., Kagita M., Yoshioka N., Tsukamoto H., Takao M., Tahara K., et al. Evaluation of the highly sensitive chemiluminescent enzyme immunoassay “Lumipulse HBsAg-HQ” for hepatitis Bvirus screening. J. Clin. Lab. Anal. 2018; 32(4): e22334. https://doi.org/10.1002/jcla.22334
- Коноплева М.В., Борисова В.Н., Соколова М.В., Фельдшерова А.А., Крымский М.A., Семененко Т.А., и др. Сравнительная характеристика антигенных свойств рекомбинантных и нативных HBs-антигенов с мутацией G145R и оценка их иммуногенности. Вопросы вирусологии. 2017; 62(4): 179–86. https://doi.org/10.18821/0507-4088-2017-62-4-179-186
- Cuestas M.L., Mathet V.L., Oubiña J.R. Specific primer sets used to amplify by PCR the hepatitis B virus overlapping S/Pol region select different viral variants. J. Viral. Hepat. 2012; 19(10): 754–6. https://doi.org/10.1111/j.1365-2893.2012.01614.x
- Zhang M., Gong Y., Osiowy C., Minuk G.Y. Rapid detection of hepatitis B virus mutations using real-time PCR and melting curve analysis. Hepatology. 2002; 36(3): 723–8. https://doi.org/10.1053/jhep.2002.35346
- Osiowy C. Sensitive Detection of HBsAg Mutants by a Gap Ligase Chain Reaction Assay. J. Clin. Microbiol. 2002; 40(7): 2566–71. https://doi.org/10.1128/JCM.40.7.2566-2571.2002
- Nainan O.V., Khristova M.L., Byun K., Xia G., Taylor P.E., Stevens C.E., et al. Genetic variation of hepatitis B surface antigen coding region among infants with chronic hepatitis B virus infection. J. Med. Virol. 2002; 68(3): 319–27. https://doi.org/10.1002/jmv.10206
- Osiowy C. Detection of HBsAg mutants. J. Med. Virol. 2006; 78(S1): S48–51. https://doi.org/10.1002/jmv.20607
- Gauthier M., Bonnaud B., Arsac M., Lavocat F., Maisetti J., Kay A., et al. Microarray for hepatitis B virus genotyping and detection of 994 mutations along the genome. J. Clin. Microbiol. 2010; 48(11): 4207–15. https://doi.org/10.1128/JCM.00344-10
- Баженов А.И., Коноплева М.В., Эльгорт Д.А., Фельдшерова А.А., Будницкая П.З., Никитина Н.И., и др. Алгоритм серологического поиска и оценка распространенности серологически значимых HBsAg-мутаций у хронических носителей вируса гепатита В. Журнал микробиологии, эпидемиологии и иммунобиологии. 2007; 6: 30–7.
- Tijssen P., Kurstak E. Highly efficient and simple methods for the preparation of peroxidase and active peroxidase-antibody conjugates ensyme immunoassays. Anal. Biochem. 1984; 136 (2): 451–7. https://doi.org/10.1016/0003-2697(84)90243-4
- Pumpens P., Grensa E., Nassal M. Molecular Epidemiology and Immunology of Hepatitis B Virus Infection. Intervirology. 2002; 45: 218–32.
- Zheng X., Weinbergerc K.M., Gehrked R., Isogawa M., Hilkene G., Kempera T., et al. Mutant hepatitis B virus surface antigens (HBsAg) are immunogenic but may have a changed specificity. Virology. 2004; 329: 454–64. https://doi.org/10.1016/j.virol.2004.08.033
- Weber B. Genetic variability of the S gene of hepatitis B virus: clinical and diagnostic impact. J. Clin. Virol. 2005; 32: 102–12. https://doi.org/10.1016/j.jcv.2004.10.008
- Norder H., Hammas B., Löfdahl S., Couroucé A.M., Magnius L.O. Comparison of the amino acid sequences of nine different serotypes of hepatitis B surface antigen and genomic classification of the corresponding hepatitis B virus strains. J. Gen. Virol. 1992; 73(5): 1201–8. https://doi.org/10.1099/0022-1317-73-5-1201
- Norder H., Couroucé A.M., Coursaget P., Echevarria J.M., Lee S.D., Mushahwar I.K., et al. Genetic diversity of hepatitis B virus strains derived worldwide: genotypes, subgenotypes, and HBsAg subtypes. Intervirology. 2004; 47(6): 289–309. https://doi.org/10.1159/000080872
- Pult I., Chouard T., Wieland S., Klemenz R., Yaniv M., Blum H.E. A hepatitis B virus mutant with a new hepatocyte nuclear factor 1 binding site emerging in transplant-transmitted fulminant hepatitis B. Hepatology. 1997; 25(6): 1507–15. https://doi.org/10.1002/hep.510250633
Supplementary files