Identification of human genes potentially involved in the pathogenesis of viral hepatitis C based on multi-network bioinformatics analysis
- 作者: Anufrieva E.V.1, Ostankova Y.V.1, Davydenko V.S.1, Schemelev A.N.1, Totolian A.A.1
-
隶属关系:
- St. Petersburg Pasteur Institute
- 期: 卷 70, 编号 3 (2025)
- 页面: 267-281
- 栏目: ORIGINAL RESEARCHES
- URL: https://journals.rcsi.science/0507-4088/article/view/310664
- DOI: https://doi.org/10.36233/0507-4088-314
- EDN: https://elibrary.ru/KFJUSV
- ID: 310664
如何引用文章
详细
Aim. The aim of this study was to search for human genes potentially involved in the pathogenesis of hepatitis C by multi-network bioinformatics linkage analysis of proteins involved in the stages of hepatitis C virus (HСV) attachment and entry.
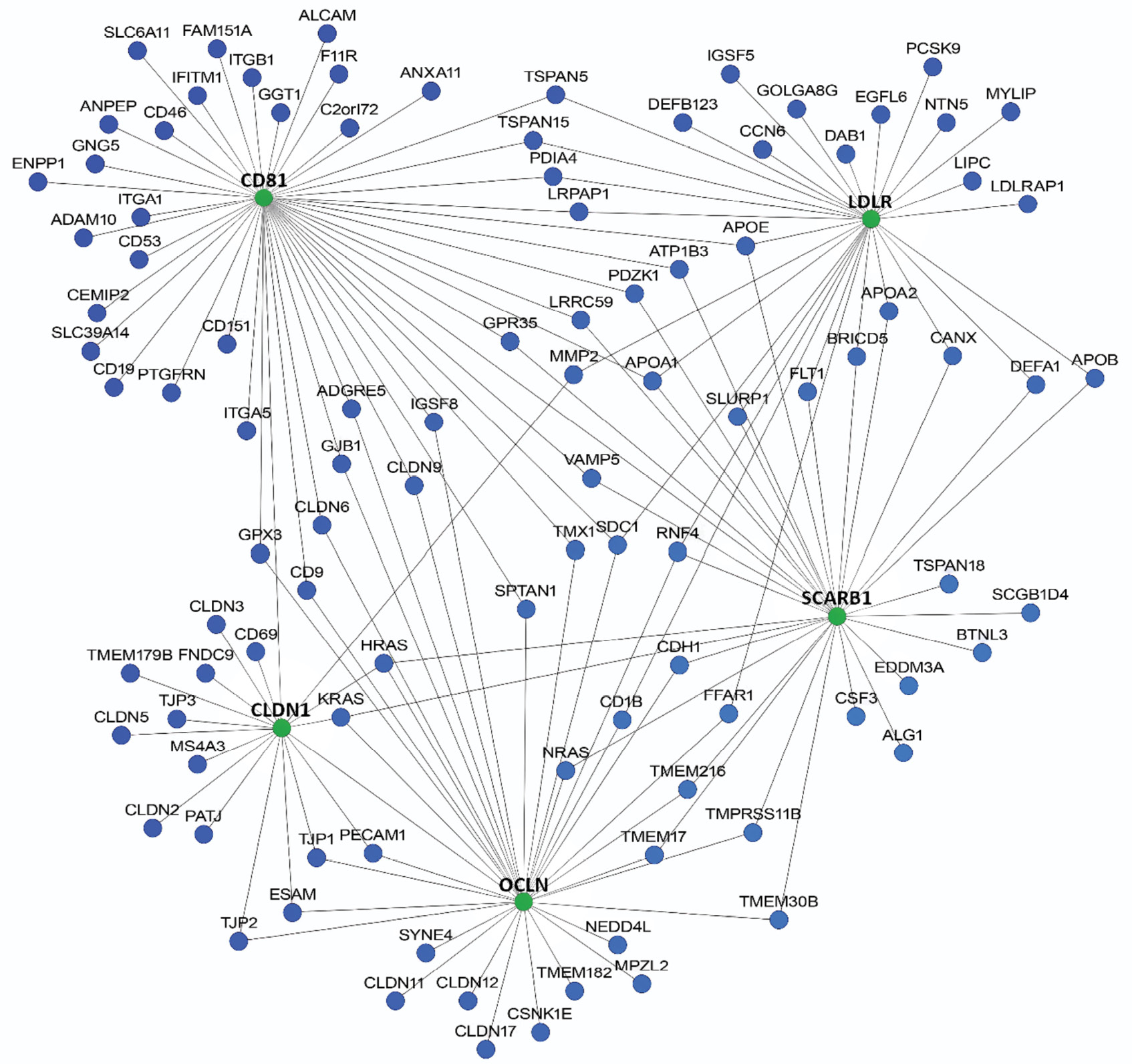
Materials and methods. A number of web applications with complementary algorithms and databases were used to analyze genetic and protein-protein networks. The following genes were used as basic genes: CD81, CLDN1, LDLR, OCLN, SCARB1, the products of which are involved in interaction with viral glycoproteins E1 and E2 at the stage of HCV attachment and penetration into the cell. Data analysis was performed, including a two-stage scoring ranking of the identified candidate genes based on their interaction with basic genes and their presence in the results of network analysis of different web resources.
Results. Candidate genes were initially identified using three web resources: HumanNet – 100 candidate genes, GeneMania – 20, STRING – 98. Based on the intersection of the three web resources, the total number of candidate genes associated with basic genes was 170. The total number of genes with a rank higher than 4 points was 35. Candidate genes were grouped into functional sets: cellular barriers and intercellular contacts (17 genes, 48.6%); lipid metabolism and lipoproteins (9 genes, 25.7%); immune response and interaction with the virus (5 genes, 14.3%); signaling pathways, proteolysis and cytoskeleton (4 genes, 11.4%). The following candidate genes potentially involved in the pathogenesis of HCV have been identified: APOA1, CLDN3, APOE, LIPC, LRPAP1, CSNK1E, APOB, CD19, CLDN6, CLDN9, ESAM, F11R, IFITM1, LDLRAP1, PCSK9, TJP1, CD9, CLDN11, CLDN17, CLDN2, CLDN5, IGSF8, MMP2, PDZK1, ADAM10, APOA2, C3, CLDN12, DAB1, GJB1, ITGB1, MYLIP, NEDD4L, PTGFRN.
Conclusion. In the future, a detailed study of the functional features and polymorphic variants of the identified genes using bioinformatics and laboratory methods can significantly expand current understanding of the involvement of human genes in the development of HCV infection and discover new targets for the development of drugs and therapeutic strategies.
作者简介
Ekaterina Anufrieva
St. Petersburg Pasteur Institute
编辑信件的主要联系方式.
Email: kate.an21@yandex.ru
ORCID iD: 0009-0002-1882-529X
Junior Researcher, Laboratory of Immunology and Virology HIV Infection, Postgraduate Student
俄罗斯联邦, St PetersburgYulia Ostankova
St. Petersburg Pasteur Institute
Email: shenna1@yandex.ru
ORCID iD: 0000-0003-2270-8897
PhD, Senior Researcher at the Laboratory of Molecular Immunology, Head of the Laboratory of Immunology and Virology HIV Infection
俄罗斯联邦, St PetersburgVladimir Davydenko
St. Petersburg Pasteur Institute
Email: vladimir_david@mail.ru
ORCID iD: 0000-0003-0078-9681
Junior Researcher, Laboratory of Immunology and Virology HIV Infection, Postgraduate Student
俄罗斯联邦, St PetersburgAlexandr Schemelev
St. Petersburg Pasteur Institute
Email: tvildorm@gmail.com
ORCID iD: 0000-0002-3139-3674
PhD, Junior Researcher, Laboratory of Immunology and Virology HIV Infection, Postgraduate Student
俄罗斯联邦, St PetersburgAreg Totolian
St. Petersburg Pasteur Institute
Email: totolian@pasteurorg.ru
ORCID iD: 0000-0003-4571-8799
Academician of the Russian Academy of Sciences, PhD, MD (Medicine), Professor, Head at the Laboratory of Molecular Immunology, Director, head Department of Immunology, First St. Petersburg State Medical University named after Academician I.P. Pavlov
俄罗斯联邦, St Petersburg参考
- Valutite D., Ostankova Y., Semenov A., Lyalina L., Totolian A. Distribution of primary resistance mutations in Saint Petersburg in patients with chronic hepatitis C. Diagnostics (Basel). 2022; 12(5): 1054. https://doi.org/10.3390/diagnostics12051054
- Guss D., Sherigar J., Rosen P., Mohanty S.R. Diagnosis and management of hepatitis C Infection in primary care settings. J. Gen. Intern. Med. 2018; 33(4): 551–7. https://doi.org/10.1007/s11606-017-4280-y
- Stanislovaitiene D., Lesauskaite V., Zaliuniene D., Smalinskiene A., Gustiene O., Zaliaduonyte-Peksiene D., et al. SCARB1 single nucleotide polymorphism (rs5888) is associated with serum lipid profile and myocardial infarction in an age- and gender-dependent manner. Lipids Health Dis. 2013; 12: 24. https://doi.org/10.1186/1476-511X-12-24
- Itakura J., Nagayama K., Enomoto N., Sakamoto N., Tazawa J., Izumi N., et al. CD81 nucleotide mutation in hepatocellular carcinoma and lack of CD81 polymorphism in patients at stages of hepatitis C virus infection. J. Med. Virol. 2001; 63(1): 22–8.
- Gerold G., Moeller R., Pietschmann T. Hepatitis C virus entry: protein interactions and fusion determinants governing productive hepatocyte invasion. Cold Spring Harb. Perspect. Med. 2020; 10(2): a036830. https://doi.org/10.1101/cshperspect.a036830
- Zeisel M.B., Felmlee D.J., Baumert T.F. Hepatitis C virus entry. Curr. Top. Microbiol. Immunol. 2013; 369: 87–112. https://doi.org/10.1007/978-3-642-27340-7_4
- Carriquí-Madroñal B., Sheldon J., Duven M., Stegmann C., Cirksena K., Wyler E., et al. The matrix metalloproteinase ADAM10 supports hepatitis C virus entry and cell-to-cell spread via its sheddase activity. PLoS Pathog. 2023; 19(11): e1011759. https://doi.org/10.1371/journal.ppat.1011759
- Yamamoto S., Fukuhara T., Ono C., Uemura K., Kawachi Y., Shiokawa M., et al. Lipoprotein receptors redundantly participate in entry of hepatitis C virus. PLoS Pathog. 2016; 12(5): e1005610. https://doi.org/10.1371/journal.ppat.1005610
- Davydenko V.S., Ostankova Yu.V., Shchemelev A.N., Anufrieva E.V., Kushnareva V.V., Totolian A.A. Identification of human genes interacting with HIV attachment receptors and potentially involved in disease pathogenesis based on multi-network bioinformatics analysis. VICh-infektsiya i immunosupressii. 2024; 16(4): 28–44. https://doi.org/10.22328/2077-9828-2024-16-4-28-44 (in Russian)
- Davydenko V.S., Ostankova Y.V., Schemelev A.N., Anufrieva E.V., Kushnareva V.V., Totolian A.A. Bioinformatically analyzed relationships between specific human genes associated with HIV attachment. Russian Journal of Infection and Immunity. 2024; 14(6): 1153–68. https://doi.org/10.15789/2220-7619-BAR-17830
- Colpitts C.C., Tsai P.L., Zeisel M.B. Hepatitis C virus entry: an intriguingly complex and highly regulated process. Int. J. Mol. Sci. 2020; 21(6): 2091. https://doi.org/10.3390/ijms21062091
- Gumbiner B.M. Breaking through the tight junction barrier. J. Cell. Biol. 1993; 123(6 Pt. 2): 1631–3. https://doi.org/10.1083/jcb.123.6.1631
- Mailly L., Baumert T.F. Hepatitis C virus infection and tight junction proteins: The ties that bind. Biochim. Biophys. Acta Biomembr. 2020; 1862(7): 183296. https://doi.org/10.1016/j.bbamem.2020.183296
- Zheng A., Yuan F., Li Y., Zhu F., Hou P., Li J., et al. Claudin-6 and claudin-9 function as additional coreceptors for hepatitis C virus. J. Virol. 2007; 81(22): 12465–71. https://doi.org/10.1128/JVI.01457-07
- Wang W., Zhou Y., Li W., Quan C., Li Y. Claudins and hepatocellular carcinoma. Biomed. Pharmacother. 2024; 171: 116109. https://doi.org/10.1016/j.biopha.2023.116109
- Park J.H., Park S., Yang J.S., Kwon O.S., Kim S., Jang S.K. Discovery of cellular proteins required for the early steps of HCV infection using integrative genomics. PLoS One. 2013; 8(4): e60333. https://doi.org/10.1371/journal.pone.0060333
- Eyre N.S., Drummer H.E., Beard M.R. The SR-BI partner PDZK1 facilitates hepatitis C virus entry. PLoS Pathog. 2010; 6(10): e1001130. https://doi.org/10.1371/journal.ppat.1001130
- Li X., Wang Q., Ai L., Cheng K. Unraveling the activation process and core driver genes of HSCs during cirrhosis by single-cell transcriptome. Exp. Biol. Med. (Maywood). 2023; 248(16): 1414–24. https://doi.org/10.1177/15353702231191109
- Rosager A.M., Sørensen M.D., Dahlrot R.H., Boldt H.B., Hansen S., Lathia J.D., et al. Expression and prognostic value of JAM-A in gliomas. J. Neurooncol. 2017; 135(1): 107–17. https://doi.org/10.1007/s11060-017-2555-0
- Brozat J.F., Brandt E.F., Stark M., Fischer P., Wirtz T.H., Flaßhove A., et al. JAM-A is a multifaceted regulator in hepatic fibrogenesis, supporting LSEC integrity and stellate cell quiescence. Liver Int. 2022; 42(5): 1185–203. https://doi.org/10.1111/liv.15187
- Abdel-Latif M.S. Plasma levels of matrix metalloproteinase (MMP)-2, MMP-9 and tumor necrosis factor-α in chronic hepatitis C virus patients. Open Microbiol. J. 2015; 9: 136–40. https://doi.org/10.2174/1874285801509010136
- Neuman M.G., Schmilovitz-Weiss H., Hilzenrat N., Bourliere M., Marcellin P., Trepo C., et al. Markers of inflammation and fibrosis in alcoholic hepatitis and viral hepatitis C. Int. J. Hepatol. 2012; 2012: 231210. https://doi.org/10.1155/2012/231210
- Samama B., Boehm N. Reelin immunoreactivity in lymphatics and liver during development and adult life. Anat. Rec. A Discov. Mol. Cell. Evol. Biol. 2005; 285(1): 595–9. https://doi.org/10.1002/ar.a.20202
- Carotti S., Perrone G., Amato M., Vespasiani Gentilucci U., Righi D., Francesconi M., et al. Reelin expression in human liver of patients with chronic hepatitis C infection. Eur. J. Histochem. 2017; 61(1): 2745. https://doi.org/10.4081/ejh.2017.2745
- Fan Y., Pionneau C., Cocozza F., Boëlle P.Y., Chardonnet S., Charrin S., et al. Differential proteomics argues against a general role for CD9, CD81 or CD63 in the sorting of proteins into extracellular vesicles. J. Extracell. Vesicles. 2023; 12(8): e12352. https://doi.org/10.1002/jev2.12352
- Li H., Wang B., Qi B., Jiang G., Qin M., Yu M. Connexin32 regulates expansion of liver cancer stem cells via the PI3K/Akt signaling pathway. Oncol. Rep. 2022; 48(3): 166. https://doi.org/10.3892/or.2022.8381
- Lee J.Y., Acosta E.G., Stoeck I.K., Long G., Hiet M.S., Mueller B., et al. Apolipoprotein E likely contributes to a maturation step of infectious hepatitis C virus particles and interacts with viral envelope glycoproteins. J. Virol. 2014; 88(21): 12422–37. https://doi.org/10.1128/JVI.01660-14
- Zeisel M.B., Felmlee D.J., Baumert T.F. Hepatitis C virus entry. Curr. Top. Microbiol. Immunol. 2013; 369: 87–112. https://doi.org/10.1007/978-3-642-27340-7_4
- Jiang J., Luo G. Apolipoprotein E but not B is required for the formation of infectious hepatitis C virus particles. J. Virol. 2009; 83(24): 12680–91. https://doi.org/10.1128/JVI.01476-09
- Labonté P., Begley S., Guévin C., Asselin M.C., Nassoury N., Mayer G., et al. PCSK9 impedes hepatitis C virus infection in vitro and modulates liver CD81 expression. Hepatology. 2009; 50(1): 17–24. https://doi.org/10.1002/hep.22911
- Malyarevskaya O.V., Namitokov A.M., Kruchinova S.V., Kosmacheva E.D. PCSK9 inhibitors: role in reducing cardiovascular diseases. Yuzhno-Rossiiskii zhurnal terapevticheskoi praktiki. 2022; 3(2): 32–40. https://doi.org/10.21886/2712-8156-2022-3-2-32-40 (in Russian)
- Desrochers G.F., Filip R., Bastianelli M., Stern T., Pezacki J.P. microRNA-27b regulates hepatic lipase enzyme LIPC and reduces triglyceride degradation during hepatitis C virus infection. J. Biol. Chem. 2022; 298(6): 101983. https://doi.org/10.1016/j.jbc.2022.101983
- Hubacek J.A., Hyatt T. ARH missense polymorphisms and plasma cholesterol levels. Clin. Chem. Lab. Med. 2004; 42(9): 989–90. https://doi.org/10.1515/CCLM.2004.200
- Lindholm D., Bornhauser B.C., Korhonen L. Mylip makes an Idol turn into regulation of LDL receptor. Cell. Mol. Life Sci. 2009; 66(21): 3399–402. https://doi.org/10.1007/s00018-009-0127-y
- Ostankova Y.V., Serikova E.N., Anufrieva E.V., Basina V.V., Mashkov I.A., Shirshova N.Yu., et al. Prognostic assessment of hepatocellular carcinoma development based on the determination of human IFNAR-1 gene polymorphism and/or its expression. Klinicheskaya laboratornaya diagnostika. 2024; 69(7): 349–57. https://doi.org/10.51620/0869-2084-2024-69-7-349-357 https://elibrary.ru/lcckcx (in Russian)
- Rehermann B. Hepatitis C virus versus innate and adaptive immune responses: a tale of coevolution and coexistence. J. Clin. Invest. 2009; 119(7): 1745–54. https://doi.org/10.1172/JCI39133
- Narayana S.K., Helbig K.J., McCartney E.M., Eyre N.S., Bull R.A., Eltahla A., et al. The interferon-induced transmembrane proteins, IFITM1, IFITM2, and IFITM3 inhibit hepatitis C virus entry. J. Biol. Chem. 2015; 290(43): 25946–59. https://doi.org/10.1074/jbc.M115.657346
- Mazumdar B., Kim H., Meyer K., Bose S.K., Di Bisceglie A.M., Ray R.B., et al. Hepatitis C virus proteins inhibit C3 complement production. J. Virol. 2012; 86(4): 2221–8. https://doi.org/10.1128/JVI.06577-11
- Assem N.M., Mohammed A.I., Barry H.M.A., El Sayed I.E.T., Elmadbouh I. Serum cystatin C is an early renal dysfunction biomarker in patients with hepatitis C virus. Egypt Liver J. 2022; 12(1): 67. https://doi.org/10.1186/s43066-022-00231-x
- Wang R.Y., Bare P., De Giorgi V., Matsuura K., Salam K.A., Grandinetti T., et al. Preferential association of hepatitis C virus with CD19+ B cells is mediated by complement system. Hepatology. 2016; 64(6): 1900–10. https://doi.org/10.1002/hep.28842
- Zhang S., Kodys K., Babcock G.J., Szabo G. CD81/CD9 tetraspanins aid plasmacytoid dendritic cells in recognition of hepatitis C virus-infected cells and induction of interferon-alpha. Hepatology. 2013; 58(3): 940–9. https://doi.org/10.1002/hep.25827
- Marquez J., Dong J., Hayashi J., Serrero G. Prostaglandin F2 receptor negative regulator (PTGFRN) expression correlates with a metastatic-like phenotype in epidermoid carcinoma, pediatric medulloblastoma, and mesothelioma. J. Cell. Biochem. 2024; 125(8): e30616. https://doi.org/10.1002/jcb.30616
- Carriquí-Madroñal B., Sheldon J., Duven M., Stegmann C., Cirksena K., Wyler E., et al. The matrix metalloproteinase ADAM10 supports hepatitis C virus entry and cell-to-cell spread via its sheddase activity. PLoS Pathog. 2023; 19(11): e1011759. https://doi.org/10.1371/journal.ppat.1011759
- Chen C., Bi Y., Chen B., He S. Nedd4L signaling contributes to carbon tetrachloride-induced liver fibrosis in female mice and is associated with enteric dysbacteriosis. Gastroenterol. Rep. (Oxf.). 2025; 13: goaf022. https://doi.org/10.1093/gastro/goaf022
- Chesarino N.M., McMichael T.M., Yount J.S. E3 ubiquitin ligase NEDD4 promotes influenza virus infection by decreasing levels of the antiviral protein IFITM3. PLoS Pathog. 2015; 11(8): e1005095. https://doi.org/10.1371/journal.ppat.1005095
- Gao P., Ma X., Yuan M., Yi Y., Liu G., Wen M., et al. E3 ligase Nedd4l promotes antiviral innate immunity by catalyzing K29-linked cysteine ubiquitination of TRAF3. Nat. Commun. 2021; 12(1): 1194. https://doi.org/10.1038/s41467-021-21456-1
- Zhou L., Fitzpatrick K., Olker C., Vitaterna M.H., Turek F.W. Casein kinase 1 epsilon and circadian misalignment impact affective behaviours in mice. Eur J Neurosci. 2022; 55(9-10): 2939-2954. https://doi.org/10.1111/ejn.15456
- Leya M., Jeong H., Yang D., Ton Nu Bao T.H., Pandeya P.R., Oh S.I., et al. Hepatocyte-specific casein kinase 1 epsilon ablation ameliorates metabolic dysfunction-associated steatohepatitis by up-regulating tumor necrosis factor receptor-associated factor 3 in mice. Am. J. Pathol. 2024; 194(11): 2106–27. https://doi.org/10.1016/j.ajpath.2024.08.003
- Harrison S.A. Steatosis and chronic hepatitis C infection: mechanisms and significance. Clin. Gastroenterol. Hepatol. 2005; 3(10 Suppl. 2): S92–6. https://doi.org/10.1016/s1542-3565(05)00706-8
补充文件