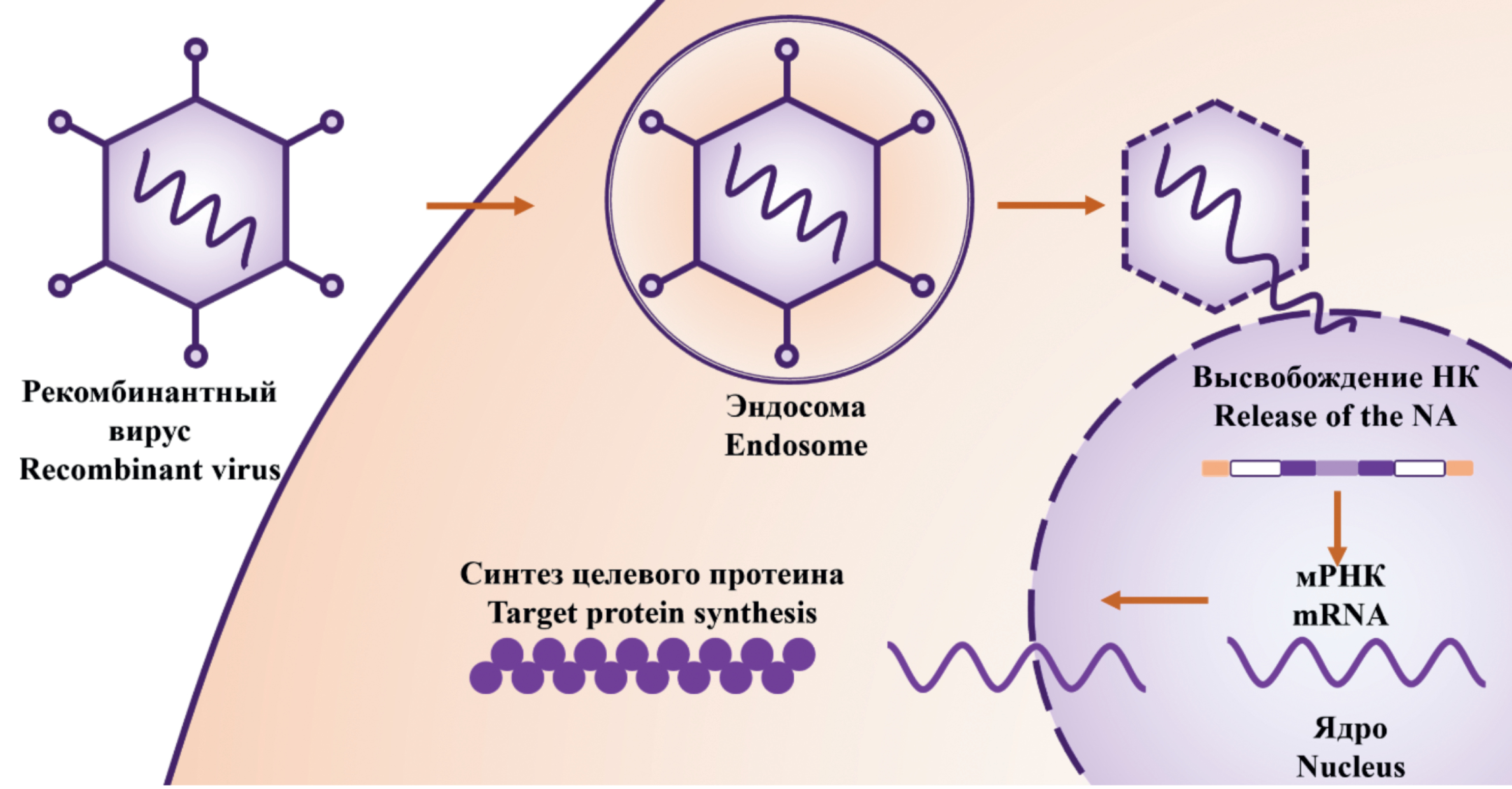
Современные подходы к конструированию и применению рекомбинантных вирусов
- Авторы: Прохорова П.В.1, Власова Н.Н.1, Южаков А.Г.1, Гулюкин А.М.1
-
Учреждения:
- ФГБНУ «Федеральный научный центр – Всероссийский научно-исследовательский институт экспериментальной ветеринарии им. К.И. Скрябина и Я.Р. Коваленко Российской академии наук»
- Выпуск: Том 70, № 5 (2025)
- Страницы: 417-430
- Раздел: ОБЗОРЫ
- URL: https://journals.rcsi.science/0507-4088/article/view/353625
- DOI: https://doi.org/10.36233/0507-4088-323
- EDN: https://elibrary.ru/cuxdyq
- ID: 353625
Цитировать
Аннотация
В обзоре описан ряд вирусных векторов и рассмотрены различные методы конструирования рекомбинантных вирусов, особое внимание уделено системе гомологичной рекомбинации и CRISPR/Cas9, описана возможность использования разных клонирующих векторов (виды плазмид, BAC). Также в обзоре представлен сравнительный анализ эффективности и безопасности применения вирусных векторов как для создания рекомбинантных вакцин, так и для получения онколитических вирусов, препаратов для генной терапии.
Полный текст
Открыть статью на сайте журналаОб авторах
Полина Владимировна Прохорова
ФГБНУ «Федеральный научный центр – Всероссийский научно-исследовательский институт экспериментальной ветеринарии им. К.И. Скрябина и Я.Р. Коваленко Российской академии наук»
Автор, ответственный за переписку.
Email: appoliproh@yandex.ru
ORCID iD: 0009-0009-7278-5541
лаборант-исследователь в Рабочей группе (отделу) по выполнению государственного задания в области инфекционной патологии животных
Россия, 109428, МоскваНаталья Никифоровна Власова
ФГБНУ «Федеральный научный центр – Всероссийский научно-исследовательский институт экспериментальной ветеринарии им. К.И. Скрябина и Я.Р. Коваленко Российской академии наук»
Email: vlanany@yandex.ru
ORCID iD: 0000-0001-8707-7710
д-р биол. наук, старший научный сотрудник, главный научный сотрудник лаборатории биохимии и молекулярной биологии
Россия, 109428, МоскваАнтон Геннадиевич Южаков
ФГБНУ «Федеральный научный центр – Всероссийский научно-исследовательский институт экспериментальной ветеринарии им. К.И. Скрябина и Я.Р. Коваленко Российской академии наук»
Email: anton_oskol@mail.ru
ORCID iD: 0000-0002-0426-9678
канд. биол. наук, заведующий лабораторией биохимии и молекулярной биологии
Россия, 109428, МоскваАлексей Михайлович Гулюкин
ФГБНУ «Федеральный научный центр – Всероссийский научно-исследовательский институт экспериментальной ветеринарии им. К.И. Скрябина и Я.Р. Коваленко Российской академии наук»
Email: admin@viev.ru
ORCID iD: 0000-0003-2160-4770
д-р ветеринар. наук, член-корр. РАН, директор
Россия, 109428, МоскваСписок литературы
- Travieso T., Li J., Mahesh S., Mello J.D.F.R.E., Blasi M. The use of viral vectors in vaccine development. NPJ Vaccines. 2022; 7(1): 75. https://doi.org/10.1038/s41541-022-00503-y
- Jogi H.R., Smaraki N., Rajak K.K., Yadav A.K., Bhatt M., Einstien C., et al. Revolutionizing Veterinary Health with Viral Vector-Based Vaccines. Indian J. Microbiol. 2024; 64(3): 867–78. https://doi.org/10.1007/s12088-024-01341-3
- Brisse M., Vrba S.M., Kirk N., Liang Y., Ly H. Emerging concepts and technologies in vaccine development. Front. Immunol. 2020; 11: 583077. https://doi.org/10.3389/fimmu.2020.583077
- Choi K.S. Newcastle disease virus vectored vaccines as bivalent or antigen delivery vaccines. Clin. Exp. Vaccine Res. 2017; 6(2): 72. https://doi.org/10.7774/cevr.2017.6.2.72
- Mendell J.R., Al-Zaidy S., Shell R., Arnold W.D., Rodino-Klapac L.R., Prior T.W., et al. Single-dose gene-replacement therapy for spinal muscular atrophy. N. Engl. J. Med. 2017; 377(18): 1713–22. https://doi.org/10.1056/NEJMoa1706198
- Ogbonmide T., Rathore R., Rangrej S.B., Hutchinson S., Lewis M., Ojilere S., et al. Gene Therapy for Spinal Muscular Atrophy (SMA): a review of current challenges and safety considerations for onasemnogene abeparvovec (zolgensma). Cureus. 2023; 15(3): e36197. https://doi.org/10.7759/cureus.36197
- Ozelo M.C., Mahlangu J., Pasi K.J., Giermasz A., Leavitt A.D., Laffan M., et al. Valoctocogene roxaparvovec gene therapy for hemophilia A. N. Engl. J. Med. 2022; 386(11): 1013–25. https://doi.org/10.1056/NEJMoa2113708
- Testa F., Bacci G., Falsini B., Iarossi G., Melillo P., Mucciolo D.P., et al. Voretigene neparvovec for inherited retinal dystrophy due to RPE65 mutations: a scoping review of eligibility and treatment challenges from clinical trials to real practice. Eye (Lond.). 2024; 38(13): 2504–15. https://doi.org/10.1038/s41433-024-03065-6
- Link E.K., Tscherne A., Sutter G., Smith E.R., Gurwith M., Chen R.T., et al. A Brighton collaboration standardized template with key considerations for a benefit/risk assessment for a viral vector vaccine based on a non-replicating modified vaccinia virus Ankara viral vector. Vaccine. 2025; 43(Pt. 1): 126521. https://doi.org/10.1016/j.vaccine.2024.126521
- Wang S., Liang B., Wang W., Li L., Feng N., Zhao Y., et al. Viral vectored vaccines: design, development, preventive and therapeutic applications in human diseases. Signal Transduct. Target Ther. 2023; 8(1): 149. https://doi.org/10.1038/s41392-023-01408-5
- Zhan W., Muhuri M., Tai P.W.L., Gao G. Vectored immunotherapeutics for infectious diseases: can rAAVs be the game changers for fighting transmissible pathogens? Front. Immunol. 2021; 12: 673699. https://doi.org/10.3389/fimmu.2021.673699
- Nascimento I.P., Leite L.C.C. Recombinant vaccines and the development of new vaccine strategies. Braz. J. Med. Biol. Res. 2012; 45(12): 1102–11. https://doi.org/10.1590/s0100-879x2012007500142
- Tukhvatulin A.I., Dolzhikova I.V., Dzharullaeva A.S., Grousova D.M., Kovyrshina A.V., Zubkova O.V., et al. Safety and immunogenicity of rAd26 and rAd5 vector-based heterologous prime-boost COVID-19 vaccine against SARS-CoV-2 in healthy adolescents: an open-label, non-randomized, multicenter, phase 1/2, dose-escalation study. Front. Immunol. 2023; 14: 1228461. https://doi.org/10.3389/fimmu.2023.1228461
- de Pinho Favaro M.T., Atienza-Garriga J., Martínez-Torró C., Parladé E., Vázquez E., Corchero J.L., et al. Recombinant vaccines in 2022: a perspective from the cell factory. Microb. Cell Fact. 2022; 21(1): 203. https://doi.org/10.1186/s12934-022-01929-8
- Valencia S., Gill R.B., Dowdell K.C., Wang Y., Hornung R., Bowman J.J., et al. Comparison of vaccination with rhesus CMV (RhCMV) soluble gB with a RhCMV replication-defective virus deleted for MHC class I immune evasion genes in a RhCMV challenge model. Vaccine. 2019; 37(2): 333–42. https://doi.org/10.1016/j.vaccine.2018.08.043
- Rasmussen T.B., Risager P.C., Fahnøe U., Friis M.B., Belsham G.J., Höper D., et al. Efficient generation of recombinant RNA viruses using targeted recombination-mediated mutagenesis of bacterial artificial chromosomes containing full-length cDNA. BMC Genomics. 2013; 14: 819. https://doi.org/10.1186/1471-2164-14-819
- Zhan X., Lee M., Xiao J., Liu F. Construction and characterization of murine cytomegaloviruses that contain transposon insertions at open reading frames m09 and M83. J. Virol. 2000; 74(16): 7411–21. https://doi.org/10.1186/1471-2164-14-819
- Urnov F.D., Miller J.C., Lee Y.L., Beausejour C.M., Rock J.M., Augustus S., et al. Highly efficient endogenous human gene correction using designed zinc-finger nucleases. Nature. 2005; 435(7042): 646–51. https://doi.org/10.1038/nature03556
- Sakuma T., Hosoi S., Woltjen K., Suzuki K.I., Kashiwagi K., Wada H., et al. Efficient TALEN construction and evaluation methods for human cell and animal applications. Genes Cells. 2013; 18(4): 315–26. https://doi.org/10.1111/gtc.12037
- Kuo L., Godeke G.J., Raamsman M.J., Masters P.S., Rottier P.J. Retargeting of coronavirus by substitution of the spike glycoprotein ectodomain: crossing the host cell species barrier. J. Virol. 2000; 74(3): 1393–406. https://doi.org/10.1128/jvi.74.3.1393-1406.2000
- Edmonds J., van Grinsven E., Prow N., Bosco-Lauth A., Brault A.C., Bowen R.A., et al. A novel bacterium-free method for generation of flavivirus infectious DNA by circular polymerase extension reaction allows accurate recapitulation of viral heterogeneity. J. Virol. 2013; 87(4): 2367–72. https://doi.org/10.1128/JVI.03162-12
- Sedova E.S., Shcherbinin D.N., Migunov A.I., Smirnov Yu.A., Logunov D.Yu., Shmarov M.M., et al. Recombinant influenza vaccines. Acta Naturae. 2012; 4(4): 17–27.
- Walsh E.P., Baron M.D., Rennie L.F., Monaghan P., Anderson J., Barrett T. Recombinant rinderpest vaccines expressing membrane-anchored proteins as genetic markers: evidence of exclusion of marker protein from the virus envelope. J. Virol. 2000; 74(21): 10165–75. https://doi.org/10.1128/jvi.74.21.10165-10175.2000
- Supotnitskiy M.V. Genotherapeutic vector systems based on viruses. Biopreparats (Biopharmaceuticals). 2011; (3): 15–26.
- Blasi M., Wescott E.C., Baker E.J., Mildenberg B., LaBranche C., Rountree W., et al. Therapeutic vaccination with IDLV-SIV-Gag results in durable viremia control in chronically SHIV-infected macaques. NPJ Vaccines. 2020; 5(1): 36. https://doi.org/10.1038/s41541-020-0186-5
- Blasi M., Negri D., LaBranche C., Alam S.M., Baker E.J., Brunner E.C., et al. IDLV-HIV-1 Env vaccination in non-human primates induces affinity maturation of antigen-specific memory B cells. Commun. Biol. 2018; 1: 134. https://doi.org/10.1038/s42003-018-0131-6
- Wang D., Wang X.W., Peng X.C., Xiang Y., Song S.B., Wang Y.Y., et al. CRISPR/Cas9 genome editing technology significantly accelerated herpes simplex virus research. Cancer Gene Ther. 2018; 25(5-6): 93–105. https://doi.org/10.1038/s41417-018-0016-3
- Chen J.S., Dagdas Y.S., Kleinstiver B.P., Welch M.M., Sousa A.A., Harrington L.B., et al. Enhanced proofreading governs CRISPR-Cas9 targeting accuracy. Nature. 2017; 550(7676): 407–10. https://doi.org/10.1038/nature24268
- Liu M., Rehman S., Tang X., Gu K., Fan Q., Chen D., et al. Methodologies for improving HDR efficiency. Front. Genet. 2018; 9: 691. https://doi.org/10.3389/fgene.2018.00691
- Tomita A., Sasanuma H., Owa T., Nakazawa Y., Shimada M., Fukuoka T., et al. Inducing multiple nicks promotes interhomolog homologous recombination to correct heterozygous mutations in somatic cells. Nat. Commun. 2023; 14(1): 5607. https://doi.org/10.1038/s41467-023-41048-5
- Nora L.C., Westmann C.A., Martins-Santana L., Alves L.F., Monteiro L.M.O., Guazzaroni M.E., et al. The art of vector engineering: towards the construction of next-generation genetic tools. Microb. Biotechnol. 2019; 12(1): 125–47. https://doi.org/10.1111/1751-7915.13318
- Julin D. Plasmid cloning vectors. In: Bell E., ed. Molecular Life Sciences. New York: Springer; 2014. https://doi.org/10.1007/978-1-4614-6436-5_86-1
- Helinski D.R. A brief history of plasmids. EcoSal Plus. 2022; 10(1): eESP00282021. https://doi.org/10.1128/ecosalplus.esp-0028-2021
- Zhou Y., Li C., Ren C., Hu J., Song C., Wang X., et al. One-step assembly of a porcine epidemic diarrhea virus infectious cDNA clone by homologous recombination in yeast: rapid manipulation of viral genome with CRISPR/Cas9 gene-editing technology. Front. Microbiol. 2022; 13: 787739. https://doi.org/10.3389/fmicb.2022.787739
- Dix T.C., Lassota A., Brauer U., Haussmann I.U., Soller M. Gap-repair recombineering for efficient retrieval of large DNA fragments from BAC clones and manipulation of large high-copy number plasmids. BMC Methods. 2024; 1(1): 11. https://doi.org/10.1186/s44330-024-00011-6
- Hao M., Tang J., Ge S., Li T., Xia N. Bacterial-artificial-chromosome-based genome editing methods and the applications in herpesvirus research. Microorganisms. 2023; 11(3): 589. https://doi.org/10.3390/microorganisms11030589.
- Chen F., Li Y.Y., Yu Y.L., Dai J., Huang J.L., Lin J. Simplified plasmid cloning with a universal MCS design and bacterial in vivo assembly. BMC Biotechnol. 2021; 21(1): 24. https://doi.org/10.1186/s12896-021-00679-6
- Li C., Wen A., Shen B., Lu J., Huang Y., Chang Y. FastCloning: a highly simplified, purification-free, sequence- and ligation-independent PCR cloning method. BMC Biotechnol. 2011; 11: 92. https://doi.org/10.1186/1472-6750-11-92
- Walhout A.J., Temple G.F., Brasch M.A., Hartley J.L., Lorson M.A., van den Heuvel S., et al. GATEWAY recombinational cloning: application to the cloning of large numbers of open reading frames or ORFeomes. Methods Enzymol. 2000; 328: 575–92. https://doi.org/10.1016/s0076-6879(00)28419-x
- Motohashi K. A simple and efficient seamless DNA cloning method using SLiCE from Escherichia coli laboratory strains and its application to SLiP site-directed mutagenesis. BMC Biotechnol. 2015; 15: 47. https://doi.org/10.1186/s12896-015-0162-8
- Engler C., Gruetzner R., Kandzia R., Marillonnet S. Golden gate shuffling: a one-pot DNA shuffling method based on type IIs restriction enzymes. PLoS One. 2009; 4(5): e5553. https://doi.org/10.1371/journal.pone.0005553
- Koskela E.V., Frey A.D. Homologous recombinatorial cloning without the creation of single-stranded ends: exonuclease and ligation-independent cloning (ELIC). Mol. Biotechnol. 2015; 57(3): 233–40. https://doi.org/10.1007/s12033-014-9817-2
- Li M.Z., Elledge S.J. Harnessing homologous recombination in vitro to generate recombinant DNA via SLIC. Nat. Methods. 2007; 4(3): 251–6. https://doi.org/10.1038/nmeth1010
- Tan L., Strong E.J., Woods K., West N.P. Homologous alignment cloning: a rapid, flexible and highly efficient general molecular cloning method. PeerJ. 2018; 6: e5146. https://doi.org/10.7717/peerj.5146
- Jeong J.Y., Yim H.S., Ryu J.Y., Lee H.S., Lee J.H., Seen D.S., et al. One-step sequence- and ligation-independent cloning as a rapid and versatile cloning method for functional genomics studies. Appl. Environ. Microbiol. 2012; 78(15): 5440–3. https://doi.org/10.1128/AEM.00844-12
- Zhu B., Cai G., Hall E.O., Freeman G.J. In-fusion assembly: seamless engineering of multidomain fusion proteins, modular vectors, and mutations. Biotechniques. 2007; 43(3): 354–9. https://doi.org/10.2144/000112536
- Zhou D., Zhou X., Bian A., Li H., Chen H., Small J.C., et al. An efficient method of directly cloning chimpanzee adenovirus as a vaccine vector. Nat. Protoc. 2010; 5(11): 1775–85. https://doi.org/10.1038/nprot.2010.134
- Davison A.J., Benkő M., Harrach B. Genetic content and evolution of adenoviruses. J. Gen. Virol. 2003; 84(Pt. 11): 2895–908. https://doi.org/10.1099/vir.0.19497-0
- Davis A.R., Wivel N.A., Palladino J.L., Tao L., Wilson J.M. Construction of adenoviral vectors. Mol. Biotechnol. 2001; 18(1): 63–70. https://doi.org/10.1385/MB:18:1:63
- Elkashif A., Alhashimi M., Sayedahmed E.E., Sambhara S., Mittal S.K. Adenoviral vector-based platforms for developing effective vaccines to combat respiratory viral infections. Clin. Transl. Immunology. 2021; 10(10): e1345. https://doi.org/10.1002/cti2.1345
- Gray G., Buchbinder S., Duerr A. Overview of STEP and Phambili trial results: two phase IIb test-of-concept studies investigating the efficacy of MRK adenovirus type 5 gag/pol/nef subtype B HIV vaccine. Curr. Opin. HIV AIDS. 2010; 5(5): 357–61. https://doi.org/10.1097/COH.0b013e32833d2d2b
- Logunov D.Y., Dolzhikova I.V., Shcheblyakov D.V., Tukhvatulin A.I., Zubkova O.V., Dzharullaeva A.S., et al. Safety and efficacy of an rAd26 and rAd5 vector-based heterologous prime-boost COVID-19 vaccine: an interim analysis of a randomised controlled phase 3 trial in Russia. Lancet. 2021; 397(10275): 671–81. https://doi.org/10.1016/S0140-6736(21)00386-X
- Wu S., Huang J., Zhang Z., Wu J., Zhang J., Hu H., et al. Safety, tolerability, and immunogenicity of an aerosolised adenovirus type-5 vector-based COVID-19 vaccine (Ad5-nCoV) in adults: preliminary report of an open-label and randomised phase 1 clinical trial. Lancet Infect. Dis. 2021; 21(12): 1654–64. https://doi.org/10.1016/S1473-3099(21)00396-0
- Zhu F.C., Li Y.H., Guan X.H., Hou L.H., Wang W.J., Li J.X., et al. Safety, tolerability, and immunogenicity of a recombinant adenovirus type-5 vectored COVID-19 vaccine: a dose-escalation, open-label, non-randomised, first-in-human trial. Lancet. 2020; 395(10240): 1845–54. https://doi.org/10.1016/S0140-6736(20)31208-3
- Xu J.W., Wang B.S., Gao P., Huang H.T., Wang F.Y., Qiu W., et al. Safety and immunogenicity of heterologous boosting with orally administered aerosolized bivalent adenovirus type-5 vectored COVID-19 vaccine and B.1.1.529 variant adenovirus type-5 vectored COVID-19 vaccine in adults 18 years and older: a randomized, double blinded, parallel controlled trial. Emerg. Microbes. Infect. 2024; 13(1): 2281355. https://doi.org/10.1080/22221751.2023.2281355
- See I., Su J.R., Lale A., Woo E.J., Guh A.Y., Shimabukuro T.T., et al. US case reports of cerebral venous sinus thrombosis with thrombocytopenia after Ad26.COV2.S vaccination, March 2 to April 21, 2021. JAMA. 2021; 325(24): 2448–56. https://doi.org/10.1001/jama.2021.7517
- Sadoff J., Gray G., Vandebosch A., Cárdenas V., Shukarev G., Grinsztejn B., et al. Safety and efficacy of single-dose Ad26.COV2.S vaccine against COVID-19. N. Engl. J. Med. 2021; 384(23): 2187–201. https://doi.org/10.1056/NEJMoa2101544
- Bos R., Rutten L., van der Lubbe J.E.M., Bakkers M.J.G., Hardenberg G., Wegmann F., et al. Ad26 vector-based COVID-19 vaccine encoding a prefusion-stabilized SARS-CoV-2 Spike immunogen induces potent humoral and cellular immune responses. NPJ Vaccines. 2020; 5: 91. https://doi.org/10.1038/s41541-020-00243-x
- Zhu F.C., Hou L.H., Li J.X., Wu S.P., Liu P., Zhang G.R., et al. Safety and immunogenicity of a novel recombinant adenovirus type-5 vector-based Ebola vaccine in healthy adults in China: preliminary report of a randomised, double-blind, placebo-controlled, phase 1 trial. Lancet. 2015; 385(9984): 2272–9. https://doi.org/10.1016/S0140-6736(15)60553-0
- Zhang Z., Zhao Z., Wang Y., Wu S., Wang B., Zhang J., et al. Comparative immunogenicity analysis of intradermal versus intramuscular immunization with a recombinant human adenovirus type 5 vaccine against Ebola virus. Front. Immunol. 2022; 13: 963049. https://doi.org/10.3389/fimmu.2022.963049
- Anywaine Z., Whitworth H., Kaleebu P., Praygod G., Shukarev G., Manno D., et al. Safety and immunogenicity of a 2-dose heterologous vaccination regimen with Ad26.ZEBOV and MVA-BN-Filo Ebola vaccines: 12-month data from a phase 1 randomized clinical trial in Uganda and Tanzania. J. Infect. Dis. 2019; 220(1): 46–56. https://doi.org/10.1016/S1473-3099(21)00128-6
- Schultz N.H., Sørvoll I.H., Michelsen A.E., Munthe L.A., Lund-Johansen F., Ahlen M.T., et al. Thrombosis and thrombocytopenia after ChAdOx1 nCoV-19 vaccination. N. Engl. J. Med. 2021; 384(22): 2124–30. https://doi.org/10.1056/NEJMoa2104882
- Greinacher A., Selleng K., Palankar R., Wesche J., Handtke S., Wolff M., et al. Insights in ChAdOx1 nCoV-19 vaccine-induced immune thrombotic thrombocytopenia. Blood. 2021; 138(22): 2256–68. https://doi.org/10.1182/blood.2021013231
- Baker A.T., Boyd R.J., Sarkar D., Teijeira-Crespo A., Chan C.K., Bates E., et al. ChAdOx1 interacts with CAR and PF4 with implications for thrombosis with thrombocytopenia syndrome. Sci. Adv. 2021; 7(49): eabl8213. https://doi.org/10.1126/sciadv.abl8213
- Ewer K., Rampling T., Venkatraman N., Bowyer G., Wright D., Lambe T., et al. A monovalent chimpanzee adenovirus Ebola vaccine boosted with MVA. N. Engl. J. Med. 2016; 374(17): 1635–46. https://doi.org/10.1056/NEJMoa1411627
- Warimwe G.M., Lorenzo G., Lopez-Gil E., Reyes-Sandoval A., Cottingham M.G., Spencer A.J., et al. Immunogenicity and efficacy of a chimpanzee adenovirus-vectored Rift Valley fever vaccine in mice. Virol. J. 2013; 10: 349. https://doi.org/10.1186/1743-422X-10-349
- Manning W.C., Paliard X., Zhou S., Pat Bland M., Lee A.Y., Hong K., et al. Genetic immunization with adeno-associated virus vectors expressing herpes simplex virus type 2 glycoproteins B and D. J. Virol. 1997; 71(10): 7960–2. https://doi.org/10.1128/JVI.71.10.7960-7962
- Derkaev A.A., Ryabova E.I., Esmagambetov I.B., Shcheblyakov D.V., Godakova S.A., Vinogradova I.D., et al. rAAV expressing recombinant neutralizing antibody for the botulinum neurotoxin type A prophylaxis. Front. Microbiol. 2022; 13: 960937. https://doi.org/10.3389/fmicb.2022.960937
- Liao G., Lau H., Liu Z., Li C., Xu Z., Qi X., et al. Single-dose rAAV5-based vaccine provides long-term protective immunity against SARS-CoV-2 and its variants. Virol. J. 2022; 19(1): 212. https://doi.org/10.1186/s12985-022-01940-w
- Pipe S.W., Leebeek F.W.G., Recht M., Key N.S., Castaman G., Miesbach W., et al. Gene therapy with etranacogene dezaparvovec for hemophilia B. N. Engl. J. Med. 2023; 388(8): 706–18. https://doi.org/10.1056/NEJMoa2211644
- Ertl H.C.J. Immunogenicity and toxicity of AAV gene therapy. Front. Immunol. 2022; 13: 975803. https://doi.org/10.3389/fimmu.2022.975803
- Pantaleo G., Janes H., Karuna S., Grant S., Ouedraogo G.L., Allen M., et al. Safety and immunogenicity of a multivalent HIV vaccine comprising envelope protein with either DNA or NYVAC vectors (HVTN 096): a phase 1b, double-blind, placebo-controlled trial. Lancet HIV. 2019; 6(11): e737–49. https://doi.org/10.1016/S2352-3018(20)30002-3
- Mastrangelo M.J., Eisenlohr L.C., Gomella L., Lattime E.C. Poxvirus vectors: orphaned and underappreciated. J. Clin. Invest. 2000; 105(8): 1031–4. https://doi.org/10.1172/JCI9819
- Draper S.J., Cottingham M.G., Gilbert S.C. Utilizing poxviral vectored vaccines for antibody induction-progress and prospects. Vaccine. 2013; 31(39): 4223–30.
- Ura T., Okuda K., Shimada M. Developments in viral vector-based vaccines. Vaccines. 2014; 2(3): 624–41. https://doi.org/10.1016/j.vaccine.2013.05.091
- Chen Z., Zhang L., Qin C., Ba L., Yi C.E., Zhang F., et al. Recombinant modified vaccinia virus Ankara expressing the spike glycoprotein of severe acute respiratory syndrome coronavirus induces protective neutralizing antibodies primarily targeting the receptor binding region. J. Virol. 2005; 79(5): 2678–88. https://doi.org/10.1128/JVI.79.5.2678-2688.2005
- Gönczöl E., Berensci K., Pincus S., Endresz V., Méric C., Paoletti E., et al. Preclinical evaluation of an ALVAC (canarypox) – human cytomegalovirus glycoprotein B vaccine candidate. Vaccine. 1995; 13(12): 1080–5. https://doi.org/10.1016/0264-410x(95)00048-6
- Naberezhnaya E.R., Soboleva A.V., Vorobyev P.O., Vadekhina V.V., Yusubalieva G.M., Isaeva I.V., et al. Interferon type I-expressing recombinant vaccinia virus as a platform for selective immunotherapy of glioblastoma and melanoma. Bulletin of Russian State Medical University. 2024; (6): 18–26. https://doi.org/10.24075/brsmu.2024.072 https://elibrary.ru/rgazed
- Кулигина Е.В., Рихтер В.А., Власов В.В. Противоопухолевый препарат на основе генно-модифицированного вируса осповакцины VV-GMCSF-LACT. Вестник Российской академии наук. 2023; 93(3): 855–64. https://doi.org/10.31857/S0869587323090098 https://elibrary.ru/ugrbdm
- Open multi-cohort study of the first phase of safety of a drug based on double recombinant vaccinia virus VV-GMCSF-Lact; 2025. Available at: https://clinicaltrials.gov/study/NCT05376527?%20cond=cancer&intr=lactaptin&rank=1
- Шакиба Я., Набережная Е.Р., Кочетков Д.В., Юсубалиева Г.М., Воробьев П.О., Чумаков П.М. и др. Сравнение онколитической активности рекомбинантных штаммов вируса осповакцины LIVP-RFP и MVA-RFP в отношении солидных опухолей. Вестник Российского государственного медицинского университета. 2023; (2): 4–12. https://doi.org/10.24075/vrgmu.2023.010
- Su D., Han L., Shi C., Li Y., Qian S., Feng Z., et al. An updated review of HSV-1 infection-associated diseases and treatment, vaccine development, and vector therapy application. Virulence. 2024; 15(1): 2425744. https://doi.org/10.1080/21505594.2024.2425744
- Ingusci S., Goins W.F., Cohen J.B., Miyagawa Y., Knipe D.M., Glorioso J.C. Next-generation replication-defective HSV vectors for delivery of large DNA payloads. Mol. Ther. 2025; 33(5): 2205–16. https://doi.org/10.1016/j.ymthe.2025.03.055
- Калиберденко В.Б., Арамян Э.Э., Зинченко М.С. Особенности онколитической виротерапии и ее применение для лечения глиобластомы. Клинический разбор в общей медицине. 2024; 5(12): 51–4. https://doi.org/10.47407/kr2024.5.12.00537
- Губанова Н.В., Гайтан А.С., Разумов И.А., Мордвинов В.А., Кривошапкин А.Л., Нетесов С.В. и др. Онколитические вирусы в терапии глиом. Молекулярная биология. 2012; 46(6): 874–86. https://elibrary.ru/pfeyjr
- Senzer N.N., Kaufman H.L., Amatruda T., Nemunaitis M., Reid T., Daniels G., et al. Phase II Clinical trial of a granulocyte-macrophage colony-stimulating factor – encoding, second-generation oncolytic herpesvirus in patients with unresectable metastatic melanoma. JCO. 2009; 27(34): 5763–71. https://doi.org/10.1200/JCO.2009.24.367
- Guide S.V., Gonzalez M.E., Bağcı I.S., Agostini B., Chen H., Feeney G., et al. Trial of Beremagene Geperpavec (B-VEC) for dystrophic epidermolysis bullosa. N. Engl. J. Med. 2022; 387(24): 2211–9. https://doi.org/10.1056/NEJMoa2206663
- Ollmann Saphire E. A vaccine against Ebola Virus. Cell. 2020; 181(1): 6. https://doi.org/10.1016/j.cell.2020.03.011
- Munis A.M., Bentley E.M., Takeuchi Y. A tool with many applications: vesicular stomatitis virus in research and medicine. Expert Opin. Biol. Ther. 2020; 20(10): 1187–201. https://doi.org/10.1080/14712598.2020.1787981
- Mire C.E., Geisbert J.B., Agans K.N., Satterfield B.A., Versteeg K.M., Fritz E.A., et al. Durability of a vesicular stomatitis virus-based Marburg virus vaccine in nonhuman primates. PLoS One. 2014; 9(4): e94355. https://doi.org/10.1371/journal.pone.0094355
- Stein D.R., Warner B.M., Soule G., Tierney K., Frost K.L., Booth S., et al. A recombinant vesicular stomatitis-based Lassa fever vaccine elicits rapid and long-term protection from lethal Lassa virus infection in guinea pigs. NPJ Vaccines. 2019; 4: 8. https://doi.org/10.1038/s41541-019-0104-x
- Hamilton A.M., Foster P.J., Ronald J.A. Evaluating nonintegrating lentiviruses as safe vectors for noninvasive reporter-based molecular imaging of multipotent mesenchymal stem cells. Hum. Gene Ther. 2018; 29(10): 1213–25. https://doi.org/10.1089/hum.2018.111
- Dong W., Kantor B. Lentiviral vectors for delivery of gene-editing systems based on CRISPR/Cas: current state and perspectives. Viruses. 2021; 13(7): 1288. https://doi.org/10.3390/v13071288
- Apolonia L. The old and the new: prospects for non-integrating lentiviral vector technology. Viruses. 2020; 12(10): 1103. https://doi.org/10.3390/v12101103
- Negri D.R.M., Rossi A., Blasi M., Michelini Z., Leone P., Chiantore M.V., et al. Simian immunodeficiency virus-Vpx for improving integrase defective lentiviral vector-based vaccines. Retrovirology. 2012; 9: 69. https://doi.org/10.1186/1742-4690-9-69
- Gallinaro A., Borghi M., Bona R., Grasso F., Calzoletti L., Palladino L., et al. Integrase defective lentiviral vector as a vaccine platform for delivering influenza antigens. Front. Immunol. 2018; 9: 171. https://doi.org/10.3389/fimmu.2018.00171
- Fontana J.M., Christos P.J., Michelini Z., Negri D., Cara A., Salvatore M. Mucosal immunization with integrase-defective lentiviral vectors protects against influenza virus challenge in mice. PLoS One. 2014; 9(5): e97270. https://doi.org/10.1371/journal.pone.0097270
- Chen Z. Parainfluenza virus 5-vectored vaccines against human and animal infectious diseases. Rev. Med. Virol. 2018; 28(2): e1965. https://doi.org/10.1002/rmv.1965
- An D., Li K., Rowe D.K., Diaz M.C.H., Griffin E.F., Beavis A.C., et al. Protection of K18-hACE2 mice and ferrets against SARS-CoV-2 challenge by a single-dose mucosal immunization with a parainfluenza virus 5-based COVID-19 vaccine. Sci. Adv. 2021; 7(27): eabi5246. https://doi.org/10.1016/j.isci.2021.103379
- Liang B., Ngwuta J.O., Herbert R., Swerczek J., Dorward D.W., Amaro-Carambot E., et al. Packaging and prefusion stabilization separately and additively increase the quantity and quality of Respiratory Syncytial Virus (RSV)-neutralizing antibodies induced by an RSV fusion protein expressed by a parainfluenza virus vector. J. Virol. 2016; 90(21): 10022–38. https://doi.org/10.1128/JVI.01196-16
- Lingemann M., Liu X., Surman S., Liang B., Herbert R., Hackenberg A.D., et al. Attenuated human parainfluenza virus type 1 expressing Ebola virus glycoprotein GP administered intranasally is immunogenic in African green monkeys. J. Virol. 2017; 91(10): e02469-16. https://doi.org/10.1128/JVI.02469-16
- Brandler S., Tangy F. Recombinant vector derived from live attenuated measles virus: potential for flavivirus vaccines. Comp. Immunol. Microbiol. Infect. Dis. 2008; 31(2-3): 271–91. https://doi.org/10.1016/j.cimid.2007.07.012
- Ebenig A., Lange M.V., Mühlebach M.D. Versatility of live-attenuated measles viruses as platform technology for recombinant vaccines. NPJ Vaccines. 2022; 7(1): 119. https://doi.org/10.1038/s41541-022-00543-4
- Desprès P., Combredet C., Frenkiel M.P., Lorin C., Brahic M., Tangy F. Live measles vaccine expressing the secreted form of the West Nile virus envelope glycoprotein protects against West Nile virus encephalitis. J. Infect. Dis. 2005; 191(2): 207–14. https://doi.org/10.1086/426824
- Rossi S.L., Comer J.E., Wang E., Azar S.R., Lawrence W.S., Plante J.A., et al. Immunogenicity and efficacy of a measles virus-vectored chikungunya vaccine in nonhuman primates. J. Infect. Dis. 2019; 220(5): 735–42. https://doi.org/10.1093/infdis/jiz202
- Nürnberger C., Bodmer B.S., Fiedler A.H., Gabriel G., Mühlebach M.D. A measles virus-based vaccine candidate mediates protection against Zika virus in an allogeneic mouse pregnancy model. J. Virol. 2019; 93(3): e01485-18. https://doi.org/10.1128/JVI.01485-18
- Fournier P., Arnold A., Wilden H., Schirrmacher V. Newcastle disease virus induces pro-inflammatory conditions and type I interferon for counter-acting Treg activity. Int. J. Oncol. 2012; 40(3): 840–50. https://doi.org/10.3892/ijo.2011.1265
- Kim S.H., Samal S. Newcastle disease virus as a vaccine vector for development of human and veterinary vaccines. Viruses. 2016; 8(7): 183. https://doi.org/10.3390/v8070183
- Kuiken T., Buijs P., Van Run P., Van Amerongen G., Koopmans M., Van Den Hoogen B. Pigeon paramyxovirus type 1 from a fatal human case induces pneumonia in experimentally infected cynomolgus macaques (Macaca fascicularis). Vet. Res. 2017; 48(1): 80. https://doi.org/10.1186/s13567-017-0486-6
- Sun W., Liu Y., Amanat F., González-Domínguez I., McCroskery S., Slamanig S., et al. A Newcastle disease virus expressing a stabilized spike protein of SARS-CoV-2 induces protective immune responses. Nat. Commun. 2021; 12(1): 6197. https://doi.org/10.1038/s41467-021-26499-y
- DiNapoli J.M., Yang L., Samal S.K., Murphy B.R., Collins P.L., Bukreyev A. Respiratory tract immunization of non-human primates with a Newcastle disease virus-vectored vaccine candidate against Ebola virus elicits a neutralizing antibody response. Vaccine. 2010; 29(1): 17–25. https://doi.org/10.1016/j.vaccine.2010.10.024
- Gonçalves M.A. Adeno-associated virus: from defective virus to effective vector. Virol. J. 2005; 2(1): 43. https://doi.org/10.1186/1743-422X-2-43
- Somia N., Verma I.M. Gene therapy: trials and tribulations. Nat. Rev. Genet. 2000; 1(2): 91–9. https://doi.org/10.1038/35038533
- Schubert M., Harmison G.G., Meier E. Primary structure of the vesicular stomatitis virus polymerase (L) gene: evidence for a high frequency of mutations. J. Virol. 1984; 51(2): 505–14. https://doi.org/10.1128/JVI.51.2.505-514.1984
- An H.Y., Kim G.N., Wu K., Kang C.Y. Genetically modified VSVNJ vector is capable of accommodating a large foreign gene insert and allows high level gene expression. Virus Res. 2013; 171(1): 168–77. https://doi.org/10.1016/j.virusres.2012.11.007
- Kalidasan V., Ng W.H., Ishola O.A., Ravichantar N., Tan J.J., Das K.T. A guide in lentiviral vector production for hard-to-transfect cells, using cardiac-derived c-kit expressing cells as a model system. Sci. Rep. 2021; 11(1): 19265. https://doi.org/10.1038/s41598-021-98657-7
- Jenkins G.M., Pagel M., Gould E.A., de A Zanotto P.M., Holmes E.C. Evolution of base composition and codon usage bias in the genus Flavivirus. J. Mol. Evol. 2001; 52: 383. https://doi.org/10.1007/s002390010168
- Duffy S., Shackelton L.A., Holmes E.C. Rates of evolutionary change in viruses: patterns and determinants. Nat. Rev. Genet. 2008; 9: 267. https://doi.org/10.1038/nrg2323
Дополнительные файлы